|
x |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGIE |
IMMUNOLOGIE |
MYCOLOGY |
PARASITOLOGY |
VIROLOGIE |
|
|
VIROLOGIE - CHAPITRE QUATRE
Stratégies de Réplication des virus à ARN
Dr Margaret Hunt
University of South Carolina School of Medicine
Columbia SC
USA
Dr Dorian McIlroy
Université de Nantes
France
|
|
EN ANGLAIS |
|
En
Español |
|
NË SHQIPTARE |
Let us know what you think
FEEDBACK |
|
SEARCH |
|
|

 |
|
Logo image © Jeffrey
Nelson, Rush University, Chicago, Illinois and
The MicrobeLibrary |
|
|
|
 Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après Virus
World
Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après Virus
World |
Réplication des virus
ARN - Généralités
a) Les virus à ARN
(Classes III, IV et V de Baltimore)
Les virus qui se répliquent via un intermédiaire en ARN
doivent obligatoirement se servir d’une ARN polymérase dépendante de l’ARN –
mais une telle activité enzymatique n’existe pas chez la cellule hôte. Tous
les virus à ARN codent donc pour une ARN polymérase dépendante de l’ARN.
Aucune protéine virale ne peut être traduite tant qu’un ARNm viral n'est pas
disponible. De ce fait, la stratégie de réplication des virus à ARN est
dépendante de la nature de l’ARN contenu dans la particule virale, car l’ARN
génomique des virus à ARN brin (+) peut servir d’ARNm directement, ce qui
n’est pas le cas de l’ARN génomique des virus à ARN brin (-) et des virus à
ARN double brin.
i. Les virus à ARN
brin (+) – Classe IV de Baltimore
Chez les virus de la classe IV de Baltimore, l’ARN génomique
est de sens positif, comme un ARNm. L’ARN génomique de ces virus peut donc
être traduit par des ribosomes cellulaires immédiatement suite à l’infection
de la cellule hôte, sans l’aide des protéines virales.
Exemples :
-
Picornaviridae (virus de la poliomyélite,
rhinovirus)
-
Togaviridae (virus de la rubéole)
-
Flaviviridae (virus de la fièvre jaune, virus de
l’hépatite C)
ii. Les virus à ARN brin (-) – Classe
V de Baltimore
Chez ces virus, l’ARN génomique est de sens négatif (c’est-à-dire,
complémentaire de l'ARNm). Il doit donc être copié en un ARN de sens positif
afin de permettre la production des protéines virales. En plus de coder pour
une ARN polymérase dépendante de l’ARN dans leur génome, les virus à ARN
brin (-) doivent également l’incorporer dans la particule virale, et la
délivrer à l’intérieur de la cellule hôte après l’entrée du virus, afin de
produire les ARNm viraux.
Exemples :
-
Orthomyxoviridae (virus de la grippe)
-
Mononegavirales
-
Rhabdoviridae (virus de la rage)
-
Paramyxoviridae (virus de la rougeole, virus des
oreillons)
iii. Les virus à ARN double brin –
Classe III de Baltimore
L’ARN génomique est double brin, et ne peut pas fonctionner
comme un ARNm. Comme les virus à ARN brin (-), les virus à ARN double brin
doivent incorporer une ARN polymérase dépendante de l’ARN à l’intérieur de
la particule virale.
Exemple :
b) Les virus à ARN qui se répliquent via un
intermédiaire en ADN
Les Retroviridae (Classe
VI de Baltimore)
L’ARN des Retroviridae, bien que de sens (+) ne fonctionne
pas en tant qu’ARNm immédiatement après l’infection, car il n’est pas libéré
dans le cytoplasme de la cellule hôte après l’entrée du virus. Au lieu de
cela, il reste associé avec certains composants de la capside, et il sert de
matrice à une transcriptase inverse, qui le copie en ADN double brin. La
transcriptase inverse n’est pas disponible dans le cytoplasme de la cellule
hôte. Les Retroviridae doivent donc coder pour cette enzyme, et l’incorporer
dans des particules virales.
|
Virus à ARN |
|
Génome |
Classe de Baltimore |
Polymérase à ARN dépendante de l’ARN à l’intérieur de la particule
virale ? |
ARN viral infectieux? |
Premier événement dans la cellule infectée |
|
ARN double brin |
III |
Oui |
Non |
Transcription |
|
ARN brin (+) |
IV |
Non |
Oui |
Traduction |
|
ARN brin (-) |
V |
Non |
Non |
Transcription |
|
RETROVIRIDAE |
|
Génome |
Classe de Baltimore |
Polymérase à ADN dépendante de l’ARN (= Transcriptase
Inverse) à l’intérieur de la particule virale ? |
ARN viral infectieux ? |
Premier événement dans la cellule infectée |
|
ARN brin (+) |
VI |
Oui |
Non |
Reverse transcription |
Le problème de la traduction
Chez les cellules eucaryotes, la machinerie moléculaire dédiée à la synthèse
protéique prend en charge des ARNm monocistroniques. C’est-à-dire que chaque
ARNm code pour un seul polypeptide – et cela pose un problème pour des virus à
ARN, qui doivent produire plusieurs types de protéine différents à partir de
leur ARN génomique. Les virus à ARN emploient plusieurs solutions différentes
à ce problème :
- Le virus transcrit plusieurs ARNm monocistroniques à partir de l’ARN
génomique (Mononegavirales)
- Le génome viral est segmenté – chaque segment code pour une protéine
virale (Orthomyxoviridae, Reoviridae)
- L’ARNm viral code pour une longue polypeptide, ou polyprotéine, qui est
ensuite clivée en de multiples différentes protéines. Toutes les protéines
virales sont donc encodées dans un seul cadre ouvert de lecture. (Picornaviridae,
Flaviviridae)
- Le transcrits primaires viraux sont subissent un épissage alternatif,
afin de produire plus d’un ARNm à partir d’un transcrit. Cette solution est
possible uniquement chez les virus qui se répliquent dans le noyau de la
cellule hôte – comme les Orthomyxoviridae.
- L’ARNm viral possède des structures secondaires spécifiques qui
permettent la fixation des ribosomes à l’interieur de la molécule, en plus,
ou à la place de l’extrémité 5’.
Taille des génomes des virus à ARN
Les virus à ARN ont des génomes de taille relativement petite,
probablement parce que l’absence d’un mécanisme de correction d’erreurs par
les ARN polymérases dépendante de l’ARN (et par les transcriptases inverses)
impose une limite supérieure à la taille d’un génome en ARN. Les génomes en
ARN les plus grands – d’environ 30 kB de longueur - se trouvent chez les
Coronaviridae.
Par conséquent, les virus à ARN codent pour un nombre limité de protéines.
Dans tous les cas, une ARN polymérase (ou une transcriptase inverse, chez
les Retroviridae) ; une ou plusieurs protéines de capside ; et (chez les
virus enveloppés) une ou plusieurs glycoprotéines de surface seront
comprises dans le génome viral.
La nécessité de coder pour ces protéines essentielles impose une limite
inférieure à la taille des génomes des virus à ARN. Chez les virus infectant
l’Homme, les génomes en ARN les plus petits – d’environ 7 kB – se trouvent
chez les Picornaviridae.
|
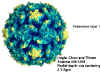 Figure 1. Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après
Virus World
Figure 1. Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après
Virus World
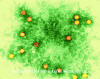 Figure 2. Le virus de la poliomyélite (grossissement 350 000x) en
microscopie électronique à coloration négative © Dennis Kunkel
Microscopy Inc., avec permission.
Figure 2. Le virus de la poliomyélite (grossissement 350 000x) en
microscopie électronique à coloration négative © Dennis Kunkel
Microscopy Inc., avec permission. |
VIRUS à ARN brin (+) – Classe IV de
Baltimore
Famille Picornaviridae
Propriétés
Les particules virales (Figure 1) sont nues, icosaédriques,
et de petite taille – d’où le nom de cette famille de virus (latin pico =
petit). L’ARN génomique est brin (+) d’une longueur de 7 ,1 à 8,9 kb.
L’extremité 5’ est fixée de façon covalente à la protéine virale VPg, et
l’extremité 3’ est polyadenylé.
Les virus type de cette famille est le virus de la poliomyélite (Figures 1,
2).
Cycle de réplication
Attachement, entrée et
décapsidation
L’attachement du virus à son récepteur cellulaire est
effectué par les protéines de capside. Chez le virus de la poliomyélite,
des acides aminés des trois protéines de l’extérieur de la capside, VP1,
VP2 et VP3, forment le site de fixation du virus à son récepteur (la PVR,
ou Polio Virus Receptor). L’interaction avec le récepteur provoque un
remaniement structural de la capside. Des domaines hydrophobes des
protéines de capside sont projetés dans la membrane plasmique de la
cellule hôte, formant un pore à travers lequel l’ARN génomique du virus
passe dans le cytoplasme. Dès son entrée dans la cellule, l’ARN viral
peut servir comme ARNm.
Synthèse des Protéines Virales
L’ARN génomique des Picornaviridae ne possède pas la «
coiffe methylé» à l’extremité 5’ qui permet la fixation des ribosomes
sur les ARNm cellulaires. Les Picornaviridae utilisent donc une
structure distincte pour l’initiation de la traduction de l’ARN viral.
Il s’agit d’une région de l’ARN génomique d’une structure secondaire en
forme de trèfle qui constitue un site interne d’entrée du ribosome
(Internal Ribosome Entry Site, ou IRES). Les facteurs d’initiation de
traduction, suivis par les ribosomes de l’hôte, se fixent sur l’IRES, ce
qui permet la traduction de la polyprotéine virale.
Souvent, les Picornaviridae inhibent la reconnaissance de la coiffé
methylé des ARNm par des facteurs d’initiation de traduction. Ainsi, les
Picornaviridae inhibent la traduction des protéines cellulaires, tout en
maintenant la traduction des protéines virales, ce qui favorise
l’expression des gènes viraux.
L’ARN viral possède une unique cadre ouvert de lecture,
qui est traduit en une longue polyprotéine. La polyprotéine est clivée
par des protéases virales (Figure 4) pendant la traduction. Certaines
des protéases virales sont actives tout en faisant partie de la
polyprotéine.
Les protéines virales libérées de la polyprotéine comprennent :
|
 Figure 3. Structure de l’ARN génomique des Picornaviridae
Figure 3. Structure de l’ARN génomique des Picornaviridae |
Réplication de l’ARN Viral
1) La polymérase à ARN virale recopie le génome brin (+)
en une copie d’ARN brin (-). Ce processus implique :
|
 Figure 4. Traduction et processing des protéines virales chez les
Picornaviridae. D’après Schaechter et al. Mechanisms of Microbial Disease
2ème Edition
Figure 4. Traduction et processing des protéines virales chez les
Picornaviridae. D’après Schaechter et al. Mechanisms of Microbial Disease
2ème Edition |
La protéine VPg sert d’amorce pour la synthèse de l’ARN
viral, ce qui explique sa présence à l’extremité 5’ de tous les ARNs
viraux.
2) L’ARN brin (-) sert de matrice pour la synthèse de
nouvelles copies brin (+) du génome viral(Figure 5). Encore une fois, la
synthèse de l’ARN viral implique la polymérase à ARN virale et la
protéine VPg.
Les nouvelles copies d’ARN brin (+) peuvent
i) Servir de matrice pour la production de copies d’ARN brin (-)
ii) Servir d’ARNm pour la production de protéines virales
iii) S’incorporer dans des capsides virales
Assemblage et libération
La réplication et l’expression de l’ARN viral se fait
dans des « usines » de production virale à proximité de vésicules
dérivées du réticulum endoplasmique. L’accumulation de protéines virales
et de capsides en cours d’assemblage au niveau de ces sites mène à la
formation de corps d’inclusion dans le cytoplasme des cellules infectées.
Dès qu’une quantité suffisante de protéines et d’ARN
viraux s’est accumulée, l’ARN viral associé avec la protéine VPg
s’associe avec les protéines de capside (VP0, VP1 et VP3) pour former
des particules immatures. La protéine VP0 est clivée en des protéines
VP2 et VP4 pour former des virions matures, infectieux. Classiquement,
on considère que la libération des particules virales se fait par lyse
de la cellule hôte, mais il semble que certains Picornaviridae sont
capables de modifier le mécanisme d’autophagie de la cellule hôte, afin
de permettre la sécrétion des particules virales.
|
 Figure 5. Réplication du génome viral chez les Picornaviridae
Figure 5. Réplication du génome viral chez les Picornaviridae |
|
A retenir à propos de la stratégie
de réplication des Picornaviridae
|
|
Cycle de réplication entièrement cytoplasmique |
|
Pas de distinction entre phases précoce et tardive |
|
Traduction initiée par la structure IRES dans l’ARN
viral |
|
Production d’une polyprotéine clivée par des
protéases virale |
|
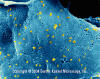 Figure 6. Particules d’un Rhabdovirus à la surface d’une cellule épithéliale
(Microscopie électronique de balayage). © Dennis Kunkel Microscopy Inc.,
avec permission.
Figure 6. Particules d’un Rhabdovirus à la surface d’une cellule épithéliale
(Microscopie électronique de balayage). © Dennis Kunkel Microscopy Inc.,
avec permission. |
VIRUS à ARN brin
(-) – Classe V de Baltimore
Ordre Mononégavirales – génomes non-segmentés
Exemples
Rhabdoviridae
Propriétés
Les particules virales sont enveloppées, avec une nucléocapside
hélicoïdale. Elles ont une forme caractéristique d’obus (Figure 7).
L’ARN génomique est brin (-) d’une longueur de 11 à 15 kb codant
pour 5 protéines.
Exemple : Le virus de la rage. Le Rhabdovirus le plus étudié est
le virus de la stomatite vésiculaire (VSV), un pathogène des porcs et d’autres
animaux d’élevage.
|
 Figure 7. Structure générale d’un Rhabdovirus
Figure 7. Structure générale d’un Rhabdovirus
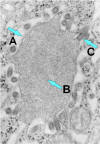 Figure 7b Bourgeonnement du virus de la rage à partir d’un
corps d’inclusion (appelé « corps Négri) vers le réticulum endoplasmique
dans une neurone. A. Corps Négri ; B. Ribonucléoprotéine virale abondante
dans le corps Négri ; Particule virale en bourgeonnement.
Image en microscopie éléctronique du CDC, Etats-Unis.
Figure 7b Bourgeonnement du virus de la rage à partir d’un
corps d’inclusion (appelé « corps Négri) vers le réticulum endoplasmique
dans une neurone. A. Corps Négri ; B. Ribonucléoprotéine virale abondante
dans le corps Négri ; Particule virale en bourgeonnement.
Image en microscopie éléctronique du CDC, Etats-Unis. |
Attachement, entrée et
décapsidation Le virus s'attache à la surface de la cellule
hôte grâce à l’interaction entre la glycoprotéine d’enveloppe du virus (G)
et son récepteur cellulaire (Figure 7).
La particule virale est ensuite internalisée par endocytose. L’acidification
de l’endosome active le domaine de fusion de la protéine G du virus, qui
provoque la fusion de l’enveloppe virale avec la membrane endosomiale. La
nucléocapside, associée avec la polymérase à ARN virale, est alors libérée
dans le cytoplasme.
Transcription et
traduction des gènes viraux Dans ce contexte, «Transcription»
fait référence à la synthèse des ARNm. Chez les Mononegavirales la
décapsidation complète de l’ARN génomique n’est pas nécessaire pour la
transcription. L'ARN polymérase du virus peut recopier l’ARN quand il est
encore associé avec la nucléocapside, car la structure de la nucléocapside
est telle que l’ARN génomique est enfilé à l’extérieur de la structure
hélicoïdale formée par la protéine de nucléocapside. Ceci est un avantage
pour le virus parce qu’il permet de protéger l’ARN génomique contre les
ribonucléases, et contre les récepteurs de la réponse immune innée de l’hôte
qui reconnaissent de l’ARN.
|
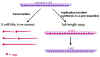 Figure 8. Transcription et réplication de l’ARN chez les Rhabdoviridae
Figure 8. Transcription et réplication de l’ARN chez les Rhabdoviridae |
Les Mononegavirales possèdent un ARNm monocistronique pour chacune des
cinq protéines codées par le virus (Figure 8). Les ARNm sont coiffés,
méthylés, et polyadénylés par la polymérase à ARN virale. Comme il s'agit
d'un virus à ARN brin (-) cytoplasmique, les activités enzymatiques pour la
synthèse et la modification de l'ARNm sont incorporées dans le virion. Les
ARNm sont traduits par les ribosomes de l’hôte. Les cinq protéines virales
sont produites en même temps, et il n’y a pas de distinction entre phases
précoce et tardive. Réplication de
l’ARN génomique La réplication de l'ARN est le processus par
lequel de nouvelles copies d'ARN génomique sont produites (figure 8). La
réplication de l'ARN a lieu dans le cytoplasme et elle est réalisée par
l'ARN polymérase virale. Le choix entre la production des ARNm et de l’ARN
brin (+) de longueur génomique est influencé par la présence de la
nucléoprotéine virale. En effet, lorsqu’une quantité suffisante de protéine
de nucléocapside est disponible, l’ARN viral nouvellement synthétisé
s’associe immédiatement avec elle. Cette association inhibe la transcription
des ARNm courts, et favorise la production d’ARN viral de longueur génomique.
Les ARNs de longueur génomiques ne sont pas coiffés, ni polyadenylés, et ne
sont pas traduits par les ribosomes de l’hôte (en particulier car ils sont
revêtus de protéine de nucléocapside).
|
 Figure 9. Transport des glycoprotéines virales du réticulum endoplasmique
vers la membrane plasmique.
Figure 9. Transport des glycoprotéines virales du réticulum endoplasmique
vers la membrane plasmique. |
Le nouveau brin (+) est copié à son tour en ARN de longueur génomique brin
(-), qui est également recouvert de protéine de nucléocapside au cours de sa
synthèse.
Les nouvelles copies d’ARN génomique brin (-) peuvent:
i. Servir de matrice pour la synthèse de copies brin (+) d’ARN génomique
ii. Servir de matrice pour la synthèse d'ARNm
iii. S’incorporer dans des virions
|
 Figure 10. Vue schématique de l’assemblage d’un Rhabdovirus par
bourgeonnement.
Figure 10. Vue schématique de l’assemblage d’un Rhabdovirus par
bourgeonnement. |
Assemblage
La particule virale comprend deux modules, l’enveloppe et la
nucléocapside.
Enveloppe:
Comme d’autres protéines transmembranaires, la protéine G des Rhabdoviridae
est transportée vers la lumière du réticulum endoplasmique, pendant sa
synthèse. Le domaine extracellulaire est glycosylé, puis la protéine G est
transportée par des vésicules à la membrane cellulaire impliqué dans le
bourgonnement des particules virales. Pour le VSV, il s’agit de la membrane
plasmique (Figure 9).
Nucléocapside:
La protéine de nucléocapside s’associe avec l’ARN génomique au cours de sa
synthèse. Les nucléocapsides quittent la cellule par bourgeonnement aux
zones de la membrane contenant les protéines virales G et M (Figure 10). La
protéine de matrice (M) interagit avec le domaine cytoplasmique de la
glycoprotéine G, et avec la nucléocapside.
|
A retenir à propos de la stratégie de réplication
des Rhabdoviridae |
| Cycle de
réplication entièrement cytoplasmique |
| Pas de
distinction entre phases précoce et tardive |
| Polymérase à
ARN dépendant de l’ARN encodée dans le génome viral et incorporée
dans le virion |
| Deux types
de transcrits distinctes – ARNm et copies d’ARN génomique |
|
 Figure 11. Un Paramyxovirus en microscopie électronique à coloration
négative © Dr Linda Stannard, Université de Cape Town (avec permission).
Figure 11. Un Paramyxovirus en microscopie électronique à coloration
négative © Dr Linda Stannard, Université de Cape Town (avec permission). |
Paramyxoviridae
Propriétés
Les particules virales (Figure 11) sont pléomorphes –
c’est-à-dire que plusieurs formes de particule virale différentes
peuvent être observées dans un même échantillon viral. La nucléocapside
de symétrie hélicoïdale est flexible, permettant son repliement à
l’intérieur de la particule (Figure 12).
L’enveloppe contient deux glycoprotéines virales : la
protéine d’attachement, et la protéine F.
-
La protéine d’attachement se fixe sur le récepteur
cellulaire. Elle peut être dotée des activités d’Hémagglutination et
de Neuraminidase (protéine HN) ; une activité d’hémagglutination
seule (protéine H); ou aucune des deux activités - elle est alors
désignée simplement comme protéine G.
-
La protéine F est responsable de la fusion
membranaire
-
L’ARN génomique est brin (-) d’une longueur
d’environ 15 kb codant pour 8 protéines.
|
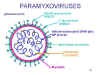 Figure 12. Structure générale d’un Paramyxovirus.
Figure 12. Structure générale d’un Paramyxovirus. |
|
Paramyxoviridae |
|
Genre
|
Glycoprotéines
|
Exemples
|
|
Sous-famille Paramyxovirinae |
|
Paramyxovirus
|
HN, F
|
Virus Parainfluenza de type 1 et 3
|
|
Rubulavirus |
HN, F |
Virus des oreillons
Virus Parainfluenza de type 2 et 4 |
|
Morbillivirus
|
H, F
|
Virus de la rougeole
|
|
Sous-famille Pneumovirinae |
|
Pneumovirus
|
G, F
|
Virus respiratoire syncytial
|
|
Metapneumovirus |
G, F |
Différents Metapneumovirus |
Hémagglutination
L’hémagglutination est facile de visualiser en laboratoire clinique et
il est une propriété utilisé en diagnostic.
Hémagglutination implique l'agglutination des globules rouges, et repose
sur la capacité d'un virus de se lier aux récepteurs des hématies. Etant
donné que les virus ont de multiples protéines de fixation par virion,
ils peuvent se lier à plus d'un globule rouge, de façon à relier des
globules rouges en un réseau. Un virus inactivé peut encore provoquer
l’hémagglutination à condition que ses protéines de fixation soient
intactes.
Si une personne développe des anticorps dirigés contre une
hémagglutinine virale, la fixation de ces anticorps sur l’hémagglutinine
empêchera sa fixation sur les globules rouges. Le sérum de cette
personne inhibera donc la réaction d'agglutination provoquée par le
virus contre lequel les anticorps sont dirigés - mais il n’inhibera pas
l’l’hémagglutination provoquée par d'autres virus. Ainsi, l’inhibition
de l’hémagglutination peut être utilisée pour déterminer quels virus
hémagglutinants ont infecté une personne par le passé.
Hémadsorption
Au cours de l’infection par certains virus enveloppés, la protéine
d’attachement virale est insérée dans la membrane plasmique de la
cellule infectée. Si la protéine d’attachement du virus possède une
activité d’hémagglutination, la cellule infectée fixera des globules
rouges - c'est ce qu'on appelle l’hémadsorption. Dans un laboratoire de
virologie clinique, l’hémadsorption peut permettre la détection de
stades précoces d’une infection en culture cellulaire, avant
l’apparition d’un effet cytopathogène prononcé.
|
 Figure 13. Vue schématique de l’attachement et entrée des Paramyxoviridae.
Figure 13. Vue schématique de l’attachement et entrée des Paramyxoviridae. |
Attachement, entrée et
décapsidation La protéine d’attachement H(N)/G reconnaît des
récepteurs à la surface des cellules, et la protéine F facilite la fusion de
l’enveloppe virale avec la membrane plasmique. Chez les Paramyxoviridae la
protéine F est capable d’induire la fusion membranaire à pH neutre, et par
conséquent, ces virus possèdent la capacité d’entrer dans la cellule par
fusion directe avec la membrane plasmique (figure 13).
Une autre conséquence de l’activité de la protéine F à pH physiologique,
est la formation des syncytia entre des cellules infectées par des
Paramyxoviridae et des cellules non-infectées voisines.
|
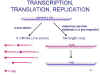 Figure 14. Transcription et réplication de l’ARN chez les Paramyxoviridae.
Figure 14. Transcription et réplication de l’ARN chez les Paramyxoviridae. |
Transcription,
traduction et réplication de l’ARN viral
Les événements à l'intérieur de la cellule infectée sont
très similaires à ceux observés au cours d’une infection par les
Rhabdoviridae (figure 14):
-
La réplication virale a lieu dans le cytoplasme
-
Comme il s'agit d'un virus ARN à brin négatif, la
polymérase à ARN est incorporée dans le virion
-
L'ARN polymérase virale utilise l’ARN associé avec la
nucléocapside comme matrice
-
La décapsidaton complète de l’ARN génomique n’est pas
nécessaire
-
Les ARNm viraux sont coiffés, méthylés et polyadénylés
par l’ARN polymérase virale, puis traduits par les ribosomes de la
cellule hôte.
-
Il n’y a pas de distinction entre les phases précoce et
tardive
Un ARN viral brin (+) de longueur génomique sert d’intermédiaire pour la
production de nouvelles copies de l’ARN génomique. Les brins (+) et (-) de
longueur génomique sont recouverts de protéine de nucléocapside pendant leur
synthèse (figure 14).
Les nouvelles copies d’ARN génomique brin (-) peuvent:
i. Servir de matrice pour la synthèse de copies brin (+) d’ARN génomique
ii. Servir de matrice pour la synthèse d'ARNm
iii. S’incorporer dans des virions
|
 Figure 15. Activation de la protéine de fusion (F) par clivage protéique.
Figure 15. Activation de la protéine de fusion (F) par clivage protéique.
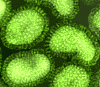 Figure 16. Le virus de la grippe A (famille Orthomyxoviridae) en
microscopie électronique à coloration négative © Dr Linda Stannard,
Université de Cape Town (avec permission).
Figure 16. Le virus de la grippe A (famille Orthomyxoviridae) en
microscopie électronique à coloration négative © Dr Linda Stannard,
Université de Cape Town (avec permission).
|
Assemblage
Les glycoprotéines virales (les protéines d’attachement et
de fusion) sont insérées dans la membrane du réticulum endoplasmique pendant
leur synthèse, et transportées vers la membrane plasmique de la cellule. La
protéine M (matrice) permet aux nucléocapsides d’interagir avec les régions
de la membrane plasmique où se trouvent les glycoprotéines virales.
Les particules virales sont libérées par bourgeonnement à
travers la membrane.
Le rôle de la neuraminidase
Chez les paramyxovirus qui la possède, la neuraminidase facilite la
libération des particules virales. Dans ce cas, l'acide sialique semble
constituer une partie importante du récepteur du virus. La neuraminidase
élimine l'acide sialique (acide neuraminique) de la surface de la cellule
infectée et des particules virales. Ainsi, les virions libérés par une
cellule infectée ne collent pas les uns aux autres ou à la cellule qu’ils
viennent de quitter par bourgeonnement (ou n'importe quelle autre cellule
infectée). Ils seront donc en mesure de diffuser à distance jusqu'à ce
qu'ils rencontrent une cellule non infectée.
La neuraminidase peut aussi faciliter l'infection car, si le virus se fixe
aux résidus d'acide sialique dans le mucus, il ne sera pas capable de se
fixer à un récepteur sur une cellule et ne pourra pas démarrer un cycle de
réplication. Par contre, si l'acide sialique dans le mucus est détruit par
l’activité de la neuraminidase, le virus sera libéré et pourra alors
atteindre un récepteur à la surface d’un cellule.
L'activation de la protéine F
La protéine F est synthétisée sous forme d’un précurseur inactif, appelé F0.
Le clivage protéolytique du précurseur F0 par des protéases cellulaires
résulte en la maturation de la protéine F en une forme (appelée F1-F2) qui
est capable de provoquer la fusion membranaire lorsque le virus se lie à une
autre cellule (figure 15). En l’absence du clivage de la protéine F0, les
particules virales libérées par une cellule infectée ne seront pas
infectieuses.
|
Quelques différences
entre les Rhabdoviridae et les Paramyxoviridae |
| |
Rhabdovirus |
Paramyxovirus |
|
Forme |
En obus |
Ronde, pléiomorphique |
| Glycoprotéines |
Une seule – possède les activités
d’attachement et de fusion membranaire |
Deux – une protéine d’attachement
et une protéine de fusion |
| pH de fusion |
Acide |
Neutre |
|
|
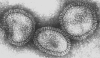 Figure 17. Le virus de la grippe A (famille Orthomyxoviridae) en
microscopie électronique à coloration négative © Dr Linda Stannard,
Université de Cape Town (avec permission).
Figure 17. Le virus de la grippe A (famille Orthomyxoviridae) en
microscopie électronique à coloration négative © Dr Linda Stannard,
Université de Cape Town (avec permission).
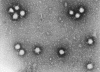 Figure 18. Un Bunyavirus en microscopie électronique à coloration
négative (d’après la base de données ICTV)
Figure 18. Un Bunyavirus en microscopie électronique à coloration
négative (d’après la base de données ICTV)
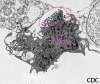 Figure 18b. Cellule Vero E6 infectée par un Arenavirus en microscopie
électronique (grossissement environ 12 000x). On peut voir des particules
libérées par bourgeonnement à la surface de la cellule. Image fournie par
Cynthia Goldsmith MS ; Division Pathologie des Maladies Infectieuses, CDC
Etats-Unis.
Figure 18b. Cellule Vero E6 infectée par un Arenavirus en microscopie
électronique (grossissement environ 12 000x). On peut voir des particules
libérées par bourgeonnement à la surface de la cellule. Image fournie par
Cynthia Goldsmith MS ; Division Pathologie des Maladies Infectieuses, CDC
Etats-Unis. |
Virus à ARN brin
(-) – génomes segmentés
Exemples
Les Orthomyxoviridae comprennent les virus de la grippe, et les Thogoto
virus, qui sont les virus des animaux transmis par les tiques.
Il existe trois groupes de virus de la grippe: A, B et
C. Les virus grippaux A et B sont les plus importants dans la maladie
humaine, et le virus de la grippe A, étant périodiquement responsable de
pandémies sévères, est le plus étudié.
Le génome est ARN brin (-), segmenté. C'est à dire que
les différents gènes qui constituent le génome viral sont codés sur des
molécules d'ARN génomique distinctes. Chez les virus de la grippe A et
B, le génome comporte 8 segments différents. Chacun de ces segments
génomiques est incorporé dans la particule virale sous forme d'une
nucléocapside hélicoïdale associée avec l'ARN polymérase virale, qui est
composée de 3 sous-unités, PA, PB1 et PB2 (Figure 19).
Les particules sont enveloppées, polymorphes (c'est à
dire qu'ils varient en forme), et possèdent deux glycoprotéines
d'enveloppe (Figure 19).
HA - Hémagglutinine - C'est la protéine d'attachement et
de fusion
NA - Neuraminidase - Elle élimine l'acide sialique de la surface du
virus et de la cellule hôte, et facilite la libération des particules
virales des cellules infectées.
|
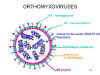 Figure 19. Structure générale d’un Orthomyxovirus
Figure 19. Structure générale d’un Orthomyxovirus |
Attachement et Entrée
Le virion s'attache à des récepteurs comportant l'acide sialique sur la
surface de la cellule et il est internalisé par endocytose. Au pH acide
de l'endosome, l'hémagglutinine subit un changement de conformation qui
provoque la fusion entre l'enveloppe virale et la membrane de
l'endolysosome. Les nucléocapsides sont libérées dans le cytoplasme.
|
 Figure 20. Transcription de l’ARNm chez les Orthomyxoviridae
Figure 20. Transcription de l’ARNm chez les Orthomyxoviridae |
Transcription et traduction de
l'ARN viral Les nucléocapsides sont transportées dans le noyau,
et la synthèse des ARNm viraux et la réplication de l'ARN viral sont
nucléaires. La réplication nucléaire est très inhabituel pour un virus à
ARN. En effet il s'agit d'un caractéristique unique des Orthomyxoviridae –
tous les autres virus à ARN se répliquent dans le cytoplasme. Le virus de
la grippe exploite un mécanisme particulier pour l'initiation de
transcription des ARNm viraux.
La protéine PB2 se fixe sur l'extrémité 5' d'un ARNm coiffé de l'hôte, qui
est ensuite clivée par l'activité d'endonucléasique de la protéine PB1 pour
produire un ARN méthylé d'une longueur d'environ 13 à 15 nucléotides. Ce
fragment d'ARN est ensuite utilisé comme amorce pour la synthèse de l'ARNm
viral (figure 20). Par conséquent, tous les ARNm de la grippe ont une courte
séquence à l'extrémité 5' dérivée d'un ARN de l'hôte. L'ARN polymérase
virale prolonge l'amorce et copie l'ARN génomique du virus en ARNm, puis
rajoute une queue poly-A à l’extrémité 3'. Huit transcrits primaires sont
produits; un transcrit par segment génomique. Les segments génomiques 7 et 8
donnent lieu à des transcrits primaires qui peuvent subir un épissage
alternatif (puisque le virus de la grippe synthétise des ARN dans le noyau,
il a accès à la machinerie d'épissage de la cellule hôte). Par exemple, le
segment 7 donne naissance à deux ARNm, l'un codant pour la protéine M1 et
l'autre pour la protéine M2. Les ARNm sont exportés dans le cytoplasme, puis
traduits en protéines virales. Les protéines transmembranaires sont
transportées vers la membrane plasmique tandis que les protéines nécessaires
à la réplication de l'ARN sont importées dans le noyau.
|
| |
Réplication de l'ARN viral
La réplication de l'ARN se produit dans le noyau à l'aide de la
polymérase virale (il n'est pas encore clair si il s'agit d'une ARN
polymérase identique à celle impliquée dans la transcription des ARNm, ou
d'une forme modifiée). Une copie exacte (sans coiffe 5', et sans queue polyA)
complémentaire de chaque segment génomique du virus est produit. Ce
transcrit brin (+) est probablement recouvert de protéine de nucléocapside
au cours de sa synthèse. Les copies brin (+) des segments génomiques sont
utilisées en tant que matrices pour la synthèse de nouvelles copies des
segments génomiques du virus. Les copies brin (-) sont également associées
avec la protéine de nucléocapside pendant leur synthèse. Elles peuvent être
utilisées
1. comme matrice pour la synthèse des ARNm viraux
2. comme matrice pour la réplication du génome viral
3. exportées vers le cytoplasme pour incorporation dans de nouvelles
particules virales.
Assemblage, Libération et
Maturation
Les virions des Orthomyxoviridae sont assemblés à la membrane plasmique.
Les nucléocapsides sont exportées hors du noyau tandis que les protéines
d'enveloppe sont transportées par l'intermédiaire de l'appareil de Golgi
vers la membrane plasmique. La protéine M1 interagit à la fois avec la
nucléocapside, et les domaines cytoplasmiques des glycoprotéines HA et NA,
et permet donc l'association des nucléocapsides avec l'enveloppe virale. Les
particules virales quittent la cellule par bourgeonnement.
La protéine NA aide le virus à quitter la cellule hôte par l'élimination
de l'acide sialique des récepteurs cellulaires, et de la surface des
particules virales. La NA peut également aider le virus à pénétrer le mucus
pour atteindre les cellules épithéliales des voies respiratoires, en lui
permettant de se dissocier de récepteurs contenant de l'acide sialique dans
le mucus. La neuraminidase n'empêche pas le virus d'infecter de nouvelles
cellules, car l'endocytose est la fusion membranaire provoquée par
l'hémagglutinine est sans doute plus rapide que la destruction du récepteur
par la neuraminidase.
La protéine HA est synthétisée sous forme d'un précurseur, HA0, qui est
incapable de provoquer la fusion membranaire. Sa maturation par clivage
protéolytique en des domaines HA1 (responsable de la fixation du virus sur
son récepteur) et HA2 (responsable de la fusion membranaire), est nécessaire
pour l'acquisition de la fonction de fusion membranaire. La protéine mature
HA1-HA2 doit subir un changement de conformation, généralement causée par
l'exposition à l'environnement acide des endosomes quand il infecte la
cellule suivante, avant qu'il ne puisse provoquer la fusion.
Le clivage du précurseur HA0 en protéines HA1-HA2 se produit pendant la
sortie du virus de la cellule, ou dans le fluide extracellulaire. En
l'absence de ce clivage, des particules virales libérées d'une cellule
infectée ne sont pas infectieuses.
Pour la plupart des virus de la grippe, l'expression des protéases qui
effectuent la maturation de la protéine HA0 est restreinte à l'appareil
respiratoire. La propagation du virus est donc limité à ce tissu.
Comparaison Paramyxoviridae /
Orthomyxoviridae
Il y a des similitudes et des différences entre les Paramyxoviridae et les
Orthomyxoviridae.
Les particules des deux types de virus sont enveloppées, et contiennent un
génome en ARN brin (-) associé avec une nucléocapside hélicoïdale. Sur le plan
clinique, les deux types de virus provoquent des infections respiratoires (ou
des infections systémiques qui sont transmises par voie respiratoire, comme la
rougeole).
Cependant, sur le plan moléculaire, les deux familles sont très différentes.
Il n'existe aucune relation sérologique entre les protéines d'attachement des
Paramyxoviridae et celles des Orthomyxoviridae, et les analyses d'homologie de
séquence des polymérases à ARN n'indiquent pas de lien phylogénétique entre les
Paramyxoviridae et les Orthomyxoviridae.
|
| |
|
Propriété
|
PARAMYXOVIRIDAE
|
ORTHOMYXOVIRIDAE
|
|
Génome
|
Monocaténaire
|
Segmenté
|
|
Réplication ARN
|
Cytoplasmique
|
Nucléaire
|
|
Besoin d'une amorce ARNm de la
cellule hôte
|
Non
|
Oui
|
|
Hémagglutinine, Neuraminidase
|
Si les deux activités sont présentes, il s'agit d'une
seule protéine (HN)
|
Les deux sont présentes, mais sur des protéines
distinctes (HA et NA)
|
|
Formation des syncytia
|
Oui (Protéine de fusion fonctionne à pH neutre)
|
Non (fusion des membranes seulement à pH acide)
|
|
 Figure 21. Visualisation en 3 dimensions de la capside d’un Reovirus. La
modélisation informatique a été effectuée à partir de données de microscopie
cryo-électronique du laboratoire de Tim Baker, Purdue University. D’après
Virus World du Dr J-Y Sgro, avec permission.
Figure 21. Visualisation en 3 dimensions de la capside d’un Reovirus. La
modélisation informatique a été effectuée à partir de données de microscopie
cryo-électronique du laboratoire de Tim Baker, Purdue University. D’après
Virus World du Dr J-Y Sgro, avec permission. |
Virus à ARN double brin
Reoviridae
La famille Reoviridae englobe plusieurs genres, y compris:
- Reovirus
- Rotavirus
- Orbivirus
- Coltivirus (nommé d'après le virus de "Colorado Tick Fever")
|
 Figure 22. Structure générale d’un Reovirus. D’après Joklik et al. dans
Zinsser Microbiology (20th Edition)
Figure 22. Structure générale d’un Reovirus. D’après Joklik et al. dans
Zinsser Microbiology (20th Edition) |
Les Reoviridae sont des virus non-enveloppés, avec une
capside de symétrie icosaédrique formée de multiples couches de protéine. En
microscopie électronique, il est possible de distinguer des particules
virales matures (Figure 23, à gauche) et des particules plus petites dites "particules
core" (Figure 23, à droite). Ces observations ont mené au modèle classique
de structure des Reoviridae selon lequel les particules virales sont
composées d'une couche interne, et d'une couche externe de protéines de
capside.
L'ARN génomique est double brin, composé de 10 à 12 segments
distinctes selon le genre auquel appartient le virus (figure 22). Les
Reovirus et les Orbivirus possède 10 segments génomiques; les Rotavirus, 11;
et les Coltivirus, 12.
Il ya des différences significatives dans le cycle de
réplication des Rotavirus par rapport à d'autre virus de famille par
Reoviridae. En raison de leur importance clinique chez l'Homme, nous allons
mettre l'accent sur les Rotavirus.
|
 Figure 23. Particules d’un Rotavirus en microscopie électronique à
coloration négative. A gauche, particule virale mature ; à droite, particule
virale "core". © Dr Linda Stannard, Université de Cape Town (avec
permission).
Figure 23. Particules d’un Rotavirus en microscopie électronique à
coloration négative. A gauche, particule virale mature ; à droite, particule
virale "core". © Dr Linda Stannard, Université de Cape Town (avec
permission). |
Rotavirus
(rota = roue (de l'apparence de virions dans en microscopie
électronique)) (figure 23)
Les analyses fines de la structure des Rotavirus ont révélé que la particule
"core" est elle-même composée de deux couches de protéine, et il est admis
actuellement que les particules des Rotavirus sont composés d'une couche
interne (T=1, composée de la protéine virale VP2), d'une couche
intermédiaire (T=13, composée de la protéine virale VP6), et d'une couche
externe (T=13, composée de la protéine virale VP7, et de la protéine des
spicules, VP4). La particule "core" est donc composée des protéines VP2 et
VP6, formant les couches interne et intermédiaire de la particule mature.
Attachement, Entrée et
Décapsidation
On ne sait toujours pas précisément ce qui se passe in vivo
lors de l'entrée des Rotavirus dans la cellule. Une protéase semble
nécessaire pour cliver la protéine de capside VP4 en deux protéines (VP5* et
VP8*) et de générer une «particule sub-virale intermédiaire» (ISVP) qui est
capable de pénétrer dans la cellule hôte. In vivo, les ISVPs sont
probablement générées par la digestion protéolytique dans le tractus
gastro-intestinal. Les produits de la protéolyse de VP4, VP5* et VP8*,
forment les protéines d'attachement du virus, qui se lient à des récepteurs
de l'hôte. Il s'agit d'un processus en de multiples étapes, avec d'abord la
fixation sur l'acide sialique de l'hôte (par VP8*), puis l'internalisation
de la particule virale par endocytose, effectuée via une interaction entre
la capside virale et les intégrines de l'hôte. La protéine VP5* provoque la
pénétration des deux couches internes de la capside virale dans le
cytoplasme.
Dans le cytoplasme, l'ARN viral est copié par l'ARN
polymérase virale tout en restant à l'intérieure de la nucléocapside
composée seulement des protéines VP2 et VP6 qui forment les couches internes
et intermédiaires de la capside virale.
|
 Figure 24. Schéma du cycle de réplication des Reoviridae.
Figure 24. Schéma du cycle de réplication des Reoviridae. |
Transcription et traduction des ARNm
virauxL'ARN génomique du virus,
étant double brin, ne fonctionne pas comme un ARNm, et donc la première
étape est de transcrire de l'ARNm à partir du génome.
Les ARNm sont produits par une ARN
polymérase virale incorporée dans le virion. L'ARN est coiffé et méthylé
par une enzymes virale, qui se trouve également à l'intérieur de la
particule virale. L'ARNm viral est ensuite extrudé à partir des pores
qui se situés aux sommets de la capside.
Les ARNm sont traduits et les protéines
virales VP2 et VP6 s'assemblent pour former une capside immature. Les
ARNm sont incorporés dans la capside immature et sont ensuite recopiés à
l'intérieur de la capside pour former des ARN double brin génomique (On
ne sait pas comment le virus s'assure que chaque particule reçoit une
copie de chacun des 11 ARNm différents) (figure 24). Les nouvelles
particules "core" se mettent à leur tour à produire des ARNm.
Assemblage et Libération
Les particules "core" incorporant l'ARN
génomique, et les enzymes virales, entourés de la couche interne (VP2)
et la couche intermédiaire (VP6) de la capside sont formés dans le
cytoplasme. Ces particules acquièrent la troisième couche de la capside
en deux étapes.
Initialement, elles acquièrent une
enveloppe par bourgeonnement dans la lumière du réticulum endoplasmique.
Ensuite, cette enveloppe est perdue en même temps que l'acquisition des
protéines de la couche externe de la capside (VP7 et VP4).
Ce mécanisme de maturation des particules virales par l'acquisition,
puis la perte d'une enveloppe est un caractéristique très étrange de les
rotavirus.
Les particules virales sont libérées par lyse cellulaire, ou par
sécrétion.
|
|
|
 Retour à la section de virologie de Microbiologie et Immunologie On-line
Retour à la section de virologie de Microbiologie et Immunologie On-line
This page last changed on
Tuesday, April 18, 2017
Page maintained by
Richard Hunt
|

 Figure 1. Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après
Virus World
Figure 1. Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après
Virus World
 Figure 4. Traduction et processing des protéines virales chez les
Picornaviridae. D’après Schaechter et al. Mechanisms of Microbial Disease
2ème Edition
Figure 4. Traduction et processing des protéines virales chez les
Picornaviridae. D’après Schaechter et al. Mechanisms of Microbial Disease
2ème Edition Figure 6. Particules d’un Rhabdovirus à la surface d’une cellule épithéliale
(Microscopie électronique de balayage). © Dennis Kunkel Microscopy Inc.,
avec permission.
Figure 6. Particules d’un Rhabdovirus à la surface d’une cellule épithéliale
(Microscopie électronique de balayage). © Dennis Kunkel Microscopy Inc.,
avec permission. Figure 7. Structure générale d’un Rhabdovirus
Figure 7. Structure générale d’un Rhabdovirus Figure 8. Transcription et réplication de l’ARN chez les Rhabdoviridae
Figure 8. Transcription et réplication de l’ARN chez les Rhabdoviridae Figure 9. Transport des glycoprotéines virales du réticulum endoplasmique
vers la membrane plasmique.
Figure 9. Transport des glycoprotéines virales du réticulum endoplasmique
vers la membrane plasmique. Figure 10. Vue schématique de l’assemblage d’un Rhabdovirus par
bourgeonnement.
Figure 10. Vue schématique de l’assemblage d’un Rhabdovirus par
bourgeonnement. Figure 11. Un Paramyxovirus en microscopie électronique à coloration
négative © Dr Linda Stannard, Université de Cape Town (avec permission).
Figure 11. Un Paramyxovirus en microscopie électronique à coloration
négative © Dr Linda Stannard, Université de Cape Town (avec permission). Figure 12. Structure générale d’un Paramyxovirus.
Figure 12. Structure générale d’un Paramyxovirus. Figure 13. Vue schématique de l’attachement et entrée des Paramyxoviridae.
Figure 13. Vue schématique de l’attachement et entrée des Paramyxoviridae. Figure 14. Transcription et réplication de l’ARN chez les Paramyxoviridae.
Figure 14. Transcription et réplication de l’ARN chez les Paramyxoviridae. Figure 15. Activation de la protéine de fusion (F) par clivage protéique.
Figure 15. Activation de la protéine de fusion (F) par clivage protéique. Figure 19. Structure générale d’un Orthomyxovirus
Figure 19. Structure générale d’un Orthomyxovirus Figure 20. Transcription de l’ARNm chez les Orthomyxoviridae
Figure 20. Transcription de l’ARNm chez les Orthomyxoviridae Figure 21. Visualisation en 3 dimensions de la capside d’un Reovirus. La
modélisation informatique a été effectuée à partir de données de microscopie
cryo-électronique du laboratoire de Tim Baker, Purdue University. D’après
Virus World du Dr J-Y Sgro, avec permission.
Figure 21. Visualisation en 3 dimensions de la capside d’un Reovirus. La
modélisation informatique a été effectuée à partir de données de microscopie
cryo-électronique du laboratoire de Tim Baker, Purdue University. D’après
Virus World du Dr J-Y Sgro, avec permission.  Figure 22. Structure générale d’un Reovirus. D’après Joklik et al. dans
Zinsser Microbiology (20th Edition)
Figure 22. Structure générale d’un Reovirus. D’après Joklik et al. dans
Zinsser Microbiology (20th Edition) Figure 23. Particules d’un Rotavirus en microscopie électronique à
coloration négative. A gauche, particule virale mature ; à droite, particule
virale "core". © Dr Linda Stannard, Université de Cape Town (avec
permission).
Figure 23. Particules d’un Rotavirus en microscopie électronique à
coloration négative. A gauche, particule virale mature ; à droite, particule
virale "core". © Dr Linda Stannard, Université de Cape Town (avec
permission). Figure 24. Schéma du cycle de réplication des Reoviridae.
Figure 24. Schéma du cycle de réplication des Reoviridae.
 Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après Virus
World
Le virus de la poliomyélite © J-Y Sgro, avec permission, d’après Virus
World




