|
|
|
|
 |
 |
|
DOENÇAS
INFECCIOSAS |
BACTERIOLOGIA |
IMUNOLOGIA |
MICOLOGIA |
PARASITOLOGIA |
VIROLOGIA |
|
|
IMUNOLOGIA –
CAPÍTULO SEIS
GENÉTICA DAS
IMUNOGLOBULINAS
Dr.
Gene Mayer
Professor Emeritus
University of South Carolina School of Medine
Tradução:
PhD. Myres Hopkins
|
|
EM INGLÊS
|
|
EM ESPANHOL |
|
SHQIP - ALBANIAN |
Dê a
sua opinião
CONTATO |
|
BUSCA |
E-MAIL
DR MYRES HOPKINS |
|
ESCOLA DE MEDICINA DA
UNIVERSIDADE DA CAROLINA DO SUL |

 |
|
|
|
OBJETIVOS
Descrever a organização e expressão das famílias de genes das
imunoglobulinas.
Explicar as origens da diversidade de anticorpos.
|
HistÓRIA
Dados de
sequenciamento de aminoácidos revelaram que uma única
região C pode estar associada com muitas regiões V diferentes.
Também foi demonstrado que um único
idiotipo
pode estar associado com diferentes regiões C (ex. IgM e IgG). Para explicar
estes dados foi sugerido que talvez as duas regiões da molécula de
imunoglobulina fossem codificadas por genes separados e que as regiões
gênicas de V e C fossem de alguma forma ligadas antes que uma molécula de
imunoglobulina fosse feita (ou seja, haveria dois genes para um
polipeptídeo). Esta foi uma idéia revolucionária mas com o advento da
recnologia do DNA recombinante, foi provado ser a correta. As cadeias
pesadas e leves das imunoglobulinas são codificadas por três famílias de
genes diferentes cada uma situada em um cromossomo diferente – uma para a
cadeia pesada e uma para cada um dos tipos de cadeia leve. Cada uma dessas
famílias de genes tem algumas regiões gênicas V e uma ou mais regiões
gênicas C. As regiões gênicas V e C não são entretanto imediatamente
adjascentes uma em relação à outra.
|
PALAVRAS CHAVE
Gene V
Gene C
Região J
Região D
Peptídeo sinal
Ativador
Promotor
Diversidade de anticorpos
Teoria da linhagem germinativa
Teoria da mutação somática
Inserções na região N
Diversidade juncional
Associação combinatorial
Multi-especificidade
Seleção clonal |
Ifamílias de genes de cadeias leves
Organização de genes da linhagem germinativa
A
organização dos genes de cadeia leve kappa e lambda na
linhagem germinativa ou em células indiferenciadas é mostrado na Figura 1.
Cadeias leves lambda
A família de genes lambda é
composta de 4 regiões gênicas C, uma para cada subtipo de cadeia lambda,
e aproximadamente 30 regiões gênicas V. Cada região gênica V está
composta de dois
exons,
um (L) que codifica para um peptídeo sinal e o outro (V) que codifica
para a maior parte da região variável. A montante (lado 5’) de cada um
dos genes C há um exon adicional chamado J (junção). Os exons L, V, J e
C estão separados por
íntrons
(sequências intervenientes não codificadoras).
Cadeias leves Kappa
A família de genes de cadeia leve
kappa contém apenas um gene de região C, uma vez que há apenas um
tipo de cadeia leve kappa. Há muitos genes de região V
(aproximadamente 250) cada um deles tem um exon para o peptídeo sinal e
um exon V. Na família de genes κ há vários exons J localizados entre os
genes V e C. Todos os exons estão separados por íntrons.
|
 Figura
1
Figura
1
Organização dos genes de cadeia leve kappa e lamda na linhagem
germinativa ou células indiferenciadas |
Rearranjo Gênico e Expressão
À medida que uma célula se diferencia em uma célula B madura que irá
fazer uma cadeia leve, ocorre um rearranjo dos vários genes (exons)
e o gene começa a ser expressado como mostrado na Figura 2.
|
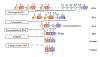 Figura
2 Figura
2
À medida que uma célula se diferencia em célula B madura que irá fazer
uma cadeia leve, ocorre o rearranjo de vários genes (exons) |
Quando a
célula se compromete a tornar-se em uma célula B fazendo uma cadeia leve, há
o rearranjo dos genes ao nível de DNA de forma que um dos genes V é levado
para próximo de uma das regiões J. Isso ocorre através de um evento
recombinacional que remove o íntron entre as regiões V e J. A seleção de
qual gene V é usado não é totalmente aleatória; há uma certa preferência
pelo uso de genes V mais próximos das regiões J. Entretanto, com o tempo
todos os genes V podem ser usados de forma que todas as combinações de genes
V e regiões J podem ser geradas.
Como
consequência deste rearranjo de DNA é que o gene se torna
transcricionalmente ativo porque um
promotor
(P), que é associado com o gene V, é levado para perto de um ativador ou
enhancer (E), que está localizado no íntron entre as regiões J e C. Quando a
transcrição se inicia a partir do promotor um pré-RNAm é feito que contém
sequências das regiões L, V J e C, assim como sequências para os íntrons
entre L e V e entre J e C (Veja Figura 2). Este pré-RNAm é processado
(splicing) no núcleo e os íntrons remanescentes são removidos. O RNAm
resultante tem os exons contíguos L, V J e C.
O RNAm é
traduzido no citoplasma e o peptídeo sinal é removido quando a proteína é
transportada pelo lúmen do retículo endoplasmático. A cadeia leve é montada
com uma cadeia pesada no retículo endoplasmático e a imunoglobulina é
secretada via rota normal das proteínas secretoras. A região V da cadeia
leve madura é codificada por sequências na região dos genes V e J e a região
J e a região C por sequências no gene C.
|
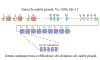 Figura 3
Figura 3
Além de vários exons J, a família de genes de cadeia pesada também
contém vários exons adicionais chamados de exons D (diversidade). Todos
os exons estão separados por íntrons |
FAMÍLIA DE GENES DE CADEIA PESADA
Organização gênica na linhagem germinativa
A
organização dos genes de cadeia pesada é mostrada na Figura 3.
Na
família de genes de cadeia pesada há muitos genes C, um para cada classe e
subclasse de imunoglobulina. Cada um dos genes C é na verdade composto por
alguns exons, um para cada domínio e outro para a região de dobradiça. Na
família de genes de cadeia pesada há muitos genes de região V, cada um
composto de uma sequência para o peptídio sinal e exon V. Além dos vários
exons J, a família de genes de cadeia pesada também contém alguns exons
adicionais chamados de exons D (diversidade). Todos os exons são separados
por íntrons como mostrado na Figura 3.
|
| |
Rearranjos gênicos e expressão
Quando uma célula se diferencia em uma célula B madura que irá fazer
uma cadeia pesada, ocorre o rearranjo de vários segmentos gênicos
(exons) e o gene começa a ser expressado como mostrado nas Figuras 4
e 5.
|
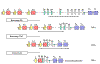 Figura 4
Figura 4
Quando a transcrição é iniciada a partir do promotor um pré-RNAm é
feito contendo as sequências para as regiões L, V, D, J Cμ e Cδ assim
como as sequências para os íntrons entre L e V, entre J e Cμ, e entre Cμ
e Cδ
 Figura
5
Figura
5
O pré-RNAm é processado no núcleo e os íntrons remanescentes, incluindo
aqueles situados entre os exons nos genes C, são removidos |
Quando
uma célula se compromete a tornar-se uma célula B fazendo uma cadeia pesada,
há dois rearranjos ao nível do DNA. Primeiro, uma das regiões D é levada
para perto de uma das regiões J e depois um dos genes V é levado para perto
da região DJ rearranjada. Isso ocorre através de dois eventos
recombinacionais que removem os íntrons entre as regiões V, D e J. Assim
como ocorre com as cadeias leves a seleção do gene V de cadeia pesada não é
totalmente aleatória mas eventualmente todos os genes V podem ser usados.
Uma
consequência destes rearranjos de DNA é que o gene se torna
transcricionalmente ativo porque um promotor (P), que está associado com o
gene V, é levado para perto de um ativador (E), que está localizado no
íntron entre as regiões J e Cmu. Quando a transcrição se
inicia a partir do promor um pré-RNAm é feito que contém sequências das
regiões L, V, D, J Cmu e Cdelta , assim
como sequências para os íntrons entre L e V, entre J e Cmu, e
entre Cmu e Cdelta (Figura 4).
O
pré-RNAm é processado (splicing) no núcleo e os íntrons remanescentes,
incluindo aqueles entre os exons nos genes C, são removidos (Veja Figura 5).
O pré-mRNA pode ser processado de duas maneiras, uma para levar VDJ para
perto do gene Cmu e a outra para levar o VDJ para perto do
gene Cdelta. Os RNAm resultantes têm os exons L, V, D, J e
Cmu ou Cdelta contíguos e irão codificar
para uma cadeia mu e uma delta, respectivamente.
Os RNAm
são traduzidos no citoplasma e o peptídeo sinal é removido à medida que a
proteína é transportada pelo lúmen do retículo endoplasmático. A cadeia
pesada é montada com uma cadeia leve no retículo endoplasmático e a
imunoglobulina é secretada via rota normal das proteínas secretoras. A
região V da cadeia pesada madura é codificada por sequências no gene V,
região D e região J e a região C por sequências no gene C.
|
 Figura
6
Figura
6
De cada lado dos exons V, J e D há sequências especiais referidas como
sequências sinais de recombinação (RSS), que funcionam na recombinação.
Cada RSS consiste de um nonâmero e um heptâmero conservado que são
separados por 12 ou 23 pares de bases. |
MECANISMOS DOS REARRANJOS DE DNA
De cada
lado dos exons V, J e D há sequências especiais referidas como sequências
sinais de recombinação (RSS), que funcionam na recombinação. Cada RSS
consiste de um nonâmero conservado e um heptâmero conservado que são
separados ou por 12 ou por 23 pares de bases (bp) como ilustrado na Figura
6. Os espaços de 12bp e 23 bp correspondem a uma ou duas voltas da hélice de
DNA.
A
recombinação só ocorre entre um sinal de 1 volta e um sinal de 2 voltas. No
caso das cadeias leves λ existe um sinal de 1 volta a montante (lado 5’) do
exon J e um sinal de 2 voltas a jusante (lado 3’) de Vlambda.
No caso das cadeias leves κ há um sinal de 1 volta a jusante (lado 3’) do
gene Vkappa e um sinal de 2 voltas a montante do exon J.
No caso das cadeias pesadas há sinais de 1 volta em cada lado do exon D e um
sinal de 2 voltas a jusante do gene V e um sinal de 2 voltas a montante do
exon J. Assim, isso garante que os eventos corretos de recombinação irão
ocorrer.
Os
eventos de recombinação resultam na remoção dos íntrons entre V e J e no
caso das cadeias leves ou entre V, D, e J no caso das cadeias pesadas. O
evento de recombinação é catalizado por duas proteínas, Rag-1 e Rag-2.
Mutações nos genes para essas proteínas resultam em uma severa doença, a
Imunodeficiência Combinada Grave (ambos os tipos de células T e B são
deficientes), já que essas proteínas e as RSS estão envolvidas na geração de
receptores para antígenos de células B e T.
|
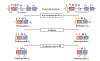
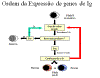 Figura 7
Ordem da expressão do gene de Ig – Cadeia pesada
Figura 7
Ordem da expressão do gene de Ig – Cadeia pesada
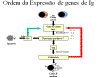 Figura
8
Figura
8
Ordem da expressão de Ig – Cadeia leve |
OrdeM DA EXPRESSÃO dos genes NAS FAMÍLIAS DE GENES DE IG
Uma
célula B individual só produz um tipo de cadeia leve e uma classe de cadeia
pesada.(Obs. A única exceção é que uma célula B madura não produz ambas as
cadeias pesadas μ e δ mas a especificidade do anticorpo é a mesma uma vez
que a mesma região VDJ é encontrada nas cadeias μ e δ). Uma vez que qualquer
célula B tem os cromossomos maternos e paternos que codificam para os genes
da imunoglobulina, deve haver uma maneira ordenada pela qual uma célula
expresse seus genes de imunoglobulina de tal forma que garanta que só um
tipo de cadeia leve e uma classe de cadeia pesada seja produzida.
A ordem
pela qual os genes de imunoglobulina são expressos em uma célula B é
mostrada na Figura 7 e 8.
Cadeia pesada
(Figura 7)
Uma célula primeiro tenta rearranjar um dos genes de cadeia pesada; em
algumas células o cromossomo materno é selecionado e em outras o cromossomo
paterno é selecionado. Se o rearranjo é bem sucedido de forma que uma cadeia
pesada é feita, então nenhum rearranjo posterior ocorre nos genes de cadeia
pesada. Se, por outro lado, a primeira tentativa de rearranjar os genes de
cadeia pesada é mal sucedido (isto é, nenhuma cadeia pesada é feita), então
a célula tenta rearranjar os genes de cadeia pesada no seu outro cromossomo.
Se a célula não for bem sucedida ao rearranjar os genes de cadeia pesada
pela segunda vez, seu destino é ser eliminada.
|
| |
Cadeia leve kappa
(Figura 8)
Quando uma célula rearranja com sucesso um gene de cadeia pesada,
ela começa então a rearranjar um dos seus genes de cadeia leve
kappa. É um evento aleatório, quer sejam selecionados genes de
cadeia leve maternos ou paternos. Se o rearranjo for mal sucedido
(isto é, não produz uma cadeia leve kappa funcional), então
ela tenta rearranjar os genes kappa do outro cromossomo. Se
uma célula rearranja um gene de cadeia leve kappa com
sucesso, ela será uma célula B que faz uma imunoglobulina com uma
cadeia leve kappa.
|
| |
Cadeia leve
lambda
(Figura 8)
Se uma célula é mal sucedida ao rearranjar ambos os
seus genes de cadeia leve kappa, ela então tenta fazer uma
cadeia leve lambda. É um evento aleatório, quer sejam
selecionados genes de cadeia leve lambda maternos ou
paternos. Se o rearranjo é mal sucedido (isto é, não produz uma
cadeia leve lambda funcional), então ela tenta rearranjar os
genes lambda no outro cromossomo. Se uma célula rearranja com
sucesso um gene de cadeia leve lambda, ela será uma célula B
que faz uma imunoglobulina com uma cadeia leve lambda.
A
sequência ordenada dos rearranjos nos genes de imunoglobulina
explica:
-
Porque uma célula B individual só pode produzir um tipo de
imunoglobulina com um tipo de cadeia pesada e um tipo de cadeia
leve.
-
Porque uma célula B individual só pode fazer anticorpos de
uma especificidade.
-
Porque há exclusão alélica em alotipos de imunoglobulina a
nível de uma molécula de imunoglobulina individual mas há
expressão co-dominante de alotipos no organismo como um todo.
|
 Figura
9
Figura
9
Idéias atuais sobre a origem da diversidade de anticorpos |
OrigEM DA DIVERSIDADE DE ANTICORPOS
Generalidades
Diversidade de anticorpos se refere à soma todal de todas as
especificidades de anticorpos que um organismo pode fazer. É istimado
que podemos fazer 107 - 108 moléculas diferentes
de anticorpos. Uma das mais importantes questões em imunologia tem sido
como podemos fazer tantas moléculas de anticorpos diferentes. As teorias
que têm tentado explicar a origem da diversidade de anticorpos caem em
duas categorias principais.
-
Teoria da linhagem germinativa
Esta teoria afirma que nós temos um gene de região V diferente para
cada anticorpo que podemos fazer.
-
Teoria da mutação somática
Esta teoria afirma que nós temos um ou poucos genes de região V e a
diversidade é gerada por mutações somáticas que ocorrem nesses
genes.
Idéias Atuais
Nosso pensamento atual é de que tanto a teoria da linhagem germinativa
como a teoria da mutação somática têm algum mérito. Acredita-se que a
diversidade de anticorpos seja gerada pelos seguintes mecanismos.
1. Um grande número de genes V
Existem:
a) 30 genes V de lambda
b) 300 genes V de kappa
c) 1000 genes V de cadeia pesada
2. Junção V-J e V-D-J
A região onde o gene V de cadeia leve e a região J ou o gene V de
cadeia pesada e as regiões D e J se unem é na terceira região
hipervariável. Uma vez que é imprevisível qual região V e quais
regiões J ou D irão se juntar, há muita diversitade que pode ser
gerada pela junção V-J e V-D-J.
3. Diversidade juncional (Inacurácias na recombinação V-J e V-D e
D-J) - (Figura 9)
|
| |
A
recombinação entre V-J e V-D-J nem sempre é perfeita e diversidade
adicional pode existir devido a êrros que ocorrem no evento de
recombinação que leva a vegião V para perto das regiões J ou D ou a
região D para perto da região J. É estimado que essas inacurácias possam
triplicar a diversidade gerada pela junção V-J e V-D-J. A diversidade
gerada por esses mecanismos ocorrem na terceira região hipervariável e
portanto, afetam diretamente o sítio de recombinação do anticorpo.
4.
Inserção na região N
Na junção entre os segmentos D e J há frequentemente uma inserção de uma
série de nucleotídeos que é catalizada pela enzima transferase terminal.
A transferase terminal cataliza a polimerização aleatória de
nucleotídeos no DNA sem a necessidade de uma fita molde. Isso leva a
mais diversidade na terceira região hipervariável.
5.
Mutação somática
Há evidência de que mutações somáticas ocorrem no gene V,
particularmente no local que codifica para a segunda região
hipervariável. Assim, mutação somática provavelmente contribui para a
diversidade de anticorpos até certo ponto.
6.
Associação combinatorial
Qualquer célula B individual tem o potencial de fazer qualquer uma das
possíveis cadeias pesadas e qualquer uma das possíveis cadeias leves.
Assim, diferentes combinações de cadeias pesadas e leves em uma célula B
individual adiciona mais diversidade.
7.
Multi-especificidade
Devido a reações cruzadas entre determinantes antigênicos de estrutura
similar um anticorpo pode frequentemente reagir com mais de um
determinante antigênico. Isso se chama multi-especificidade.
Multi-especificidade também contribui para a diversidade de anticorpos.
Um
exemplo de como estes mecanismos podem gerar uma grande quantidade de
diversidade é ilustrado abaixo:
|
| |
|
|
Receptor de Célula B (Imunoglobulina) |
|
Pesada |
Kappa |
|
Segmentos de gene V |
1000 |
300 |
|
Segmentos de gene D |
15 |
- |
|
Segmentos de gene J |
4 |
4 |
|
Inserção na região N |
++ |
- |
|
Diversidade juncional |
+++ |
+ |
|
Mutação somática |
+ |
+ |
|
Associação combinatorial |
V x D x J
1000 X 15 X 4 |
V x J
300 x 4 |
|
Total |
6 x 104 |
1.2 x 103 |
|
|
|
|
Associação combinatorial |
7.2 x 107 |
|
| |
Estes cálculos não levam em consideração as contribuições das
cadeias leves lambda, mutação somática, diversidade
juncional, inserções na região N ou multi-especificidade.
O
processo de rearranjo gênico das cadeias pesadas e leves e a
associação combinatorial dessas cadeias ocorre durante o
desenvolvimento da célula B e é independente de antígeno.
Clones de células B expressando todas as possíveis especificidades
de anticorpos são produzidos durante o desenvolvimento e antígeno
simplesmente seleciona aqueles clones que têm o receptor apropriado.
Os clones selecionados são então ativados, proliferam e se
diferenciam em plasmócitos secretores de anticorpos.
RECEPTOR DE CÉLULA T PARA ANTÍGENO
Células T também têm um receptor para antígeno em suas superfícies.
Este receptor não é uma molécula de imunoglobulina mas é composto de
duas cadeias polipeptídicas diferentes que têm regiões contantes e
variáveis análogas às das imunoglobulinas. A diversidade no receptor
de célula T também é gerada da mesma maneira como foi descrita para
a diversidade de anticorpos (ex. por junção VJ e VDJ dos
segmentos de genes e associação combinatorial). Entretanto, nenhuma
mutação somática tem sido observada em células T.
|
|


|
 Voltar
à Seção Imunologia do Microbiologia e Imunologia Online
Voltar
à Seção Imunologia do Microbiologia e Imunologia Online
Esta página foi modificada em
Página mantida por
Richard Hunt
|
