|
x |
x |
|
 |
 |
|
DOENÇAS
INFECCIOSAS |
BACTERIOLOGIA |
IMUNOLOGIA |
MICOLOGIA |
PARASITOLOGIA |
VIROLOGIA |
|
|
BACTERIOLOGIA – CAPÍTULO NOVE
MECANISMOS
GENÉTICOS REGULATÓRIOS
Dr Gene Mayer
Emeritus Professor
University of South Carolina School of Medicine
Tradução: Dr.
Myres Hopkins
|
|
INGLÊS |
|
ESPANOL |
|
ALBANES |
|
|
|
|
Dê a
sua opinião
CONTATO |
|
BUSCA |

 |
|
Logo image © Jeffrey
Nelson, Rush University, Chicago, Illinois and
The MicrobeLibrary |
APRENDA PLUGADO |
|
OBJETIVOS
Discutir a estrutura e transcrição de genes bacterianos
Descrever os mecanismos moleculares que a bactéria usa para regular a
atividade gênica
Comparar e diferenciar Operons induzíveis e repressíveis
Descrever os mecanismos moleculares envolvidos na repressão catabólica e
atenuação
Discutir maneiras que a bactéria usa para regular a atividade enzimática |
REGULAÇÃO DA EXPRESSÃO GÊNICA
Bactérias não fazem todas
as proteínas que são capazes de fazer toda a hora. Ao invés disso, elas se
adaptam ao ambiente e fazem somente aqueles produtos gênicos que são essenciais
para a sobrevivência em um ambiente em particular. Por exemplo, bactérias não
sintetizam as enzimas necessárias para fazer triptófano quando tem uma fonte
abundante de triptófano no ambiente. Entretanto, quando o triptófano está
ausente do meio ambiente as enzimas são feitas. Do mesmo modo, só porque uma
bactéria tem o gene de resistência a um antibiótico isso não quer dizer que o
gene irá se expressar. O gene de resistência vai ser expresso somente quando o
antibiótico estiver presente no meio.
Bactérias normalmente controlam a expressão gênica pela regulação do nível de
transcrição do RNAm. Em bactérias, genes com função relacionadas são geralmente
localizados adjascentes um ao outro e eles são regulados de forma coordenada (i.e.
quando um é expressado, todos os outros são também expressados). A regulação
coordenadas de conjuntos de genes é conseguida pela regulação da produção de um
RNAm policistrônico (i.e. um RNAm grande contendo a informação de vários
genes). Assim, bactérias são capazes de "perceber" o seu ambiente e expressar o
conjunto de genes apropriados para aquele ambiente através da regulação da
transcrição daqueles genes. |
|
PALAVRAS-CHAVES
Expressão gênica coordenada
RNAm policistrônico
Promotor
Operon
Operon induzível
Indutor
Gene estrutural
Gene regulatório
Repressor
Operador
Controle negativo
Repressão catabólica
Proteína CAP
Controle positivo
Operon repressível
Co-repressor
Apo-repressor
Atenuação
Região lider
Inibição por retroalimentação (feedback)
Modificação epigenética
 Figura 1
Figura 1
O operon lactose
 Figura 2 Transcrição dos genes lac na presença e ausência da
glucose
Figura 2 Transcrição dos genes lac na presença e ausência da
glucose |
Genes
induzíveis – O modêlo do operon
Definição
Um gene induzível é um gene que é expresso na presença de uma substância (um
indutor) no ambiente. Esta substância pode controlar a expressão de um ou
mais genes (genes estruturais) envolvidos no metabolismo daquela substância.
Por exemplo, lactose induz a expressão dos genes lac que estão
envolvidos no metabolismo da lactose. E certos antibióricos podem induzir a
expressão de um gene que leva à resistência àquele antibiótico.
Indução é comum em vias
metabólicas que levam ao catabolismo de uma substância e o indutor é
normalmente o substrato da via.
Operon Lactose
Genes estruturais
O operon lactose (figura 1) contém três genes estruturais que codificam para
enzimas envolvidas no metabolismo da lactose.
-
O gene lac z
codifica para a
β-galactosidase,
uma enzima que degrada lactose a glucose e galactose
-
O gene lac y
codifica para uma permease, que está envolvida na internalização da
lactose
-
O gene
lac a
gene codifica para uma galactose transacetilase.
Esses genes são
transcritos a partir de um promotor comum em um RNAm policistrônico, que é
traduzido para produzir as três enzimas.
Gene regulatório
A espressão dos genes estruturais é influenciada não somente pela presença
ou ausência do indutor, como também é controlada por um gene regulatório
específico. O gene regulatório pode estar próximo ou distante dos genes que
estão regulados. O gene regulatório codifica para um produto protêico
específico chamado de REPRESSOR
Operador
O repressor age pela ligação a uma região específica do DNA chamada de
operador, que está adjascente aos genes estruturais que estão sendo
regulados. Os genes estruturais juntamente com a região do operador e o
promotor são chamados em conjunto de OPERON. Entretanto, a ligação do
repressor ao operador é impedida pelo indutor e o indutor pode também
remover o repressor que já se ligou ao operador. Assim, na presença do
indutor o repressor é inativo e não liga ao operador, resultando na
transcrição dos genes estruturais. Contrariamente, na ausência do indutor o
repressor é ativo e liga ao operador, resultando na inibição da transcrição
dos genes estruturais. Esse tipo de controle é chamado de CONTROLE NEGATIVO,
visto que a função do produto do gene regulatório (repressor) é de desligar
a transcrição dos genes estruturais
Indutor
A transcrição dos genes lac é influenciada pela presença ou ausência de um
indutor (lactose ou outros
β-galactosídeos)
(Figura 2).
|
ex. |
+ indutor |
expressão |
|
|
- indutor |
Ausência de
expressão |
|
|
|
 Figura
3 Repressão catabólica Figura
3 Repressão catabólica
 Figura 4 Efeito da glucose na expressão de proteínas codificadas pelo
operon lac
Figura 4 Efeito da glucose na expressão de proteínas codificadas pelo
operon lac
|
 Figura 5 Efeito da glucose na expressão de proteínas codificadas pelo
operon lac
Figura 5 Efeito da glucose na expressão de proteínas codificadas pelo
operon lac |
Repressão catabólica (Efeito Glucose)
Muitos operons
induzíveis são controlados não somente por seus indutores respectivos,
como também são controlados pelo nível de glusoce no ambiente. A
habilidade da glucose de controlar a expressão de uma variedade de
operons induzíveis é chamada de REPRESSÃO CATABÓLICA. Isso é ilustrado
na Figura 3.
A repressão
catabólica é geralmente vista naqueles operons que estão envolvidos na
degradação de compostos usados como fonte de energia. Uma vez que
glucose é a fonte de energia preferida em bactéria, a habilidade da
glucose de regular a expressão de outros operons garante que a bactéria
utilize a glucose antes de qualquer outra fonte de carbono como fonte de
energia.
Mecanismo
Há uma relação inversa entre os níveis de glucose e AMP cíclico (AMPc)
em bactéria. Quando os níveis de glucose são elevados os níveis de AMPc
são baixos e quando os níveis de glucose são baixos os níveis de AMPc
são altos. Essa relação existe porque o transporte de glucose para
dentro da célula inibe a enzima adenil-ciclase, que produz AMPc. Na
célula bacteriana AMPc se liga a uma proteína de ligação ao AMPc chamada
CAP ou CRP. O complexo APMc-CAP (mas não a proteína livre CAP), se liga
a um sítio nos promotores de operons sensíveis à repressão catabólica. A
ligação do complexo resulta em um promotor mais eficiente e assim em
mais iniciações de transcrição daquele promotor, como ilustrado nas
Figuras 4 e 5. Visto que o papel do complexo CAP-AMPc é de ligar a
transcrição, este tipo de controle é dito CONTROLE POSITIVO. As
consequências deste tipo de controle é que para conseguir a expressão
máxima de um operon sensível à repressão catabólica, a glucose precisa
estar presente. Se ambos estão presentes, o operon não vai ser expresso
em sua plenitute até que a glucose seja metabolizada. Obviamente,
nenhuma expressão do operon irá ocorrer a menos que o indutor esteja
presente.
|
 Figura 6
Figura 6
O operon triptófano
 Figura 7 O efeito do triptófano na expressão do operon trip
Figura 7 O efeito do triptófano na expressão do operon trip |
Genes
repressíveis – O modelo do operon
Definição
Genes repressíveis são aqueles em que a presença de uma substância (um
co-repressor) no ambiente desliga a expressão daqueles genes (genes
estruturais) envolvidos no metabolismo daquela substância.
ex., Triptófano
reprime a expressão dos genes trp.
Repressão é comum em
vias metabólicas que levam à biossíntese de uma substância e o co-repressor
é normalmente o produto final da via que está sendo regulada.
Operon
triptófano
Genes estruturais
O operon triptófano (figura 6) contém cinco genes estruturais que codificam
para enzimas envolvidas na síntese do triptófano. Esses genes são
transcritos a partir de um promotor comum em um RNAm policistrônico, que é
traduzido para produzir as cinco enzimas.
Genes
regulatórios
A expressão dos genes estruturais é não somente influenciada pela presença
do co-repressor, como também é controlada por um gene regulatório
específico. O gene regulatório pode estar próximo ou distante dos genes que
estão sendo regulados. O gene regulatório codifica para um produto protêico
específico chamado de REPRESSOR (às vezes chamado de apo-repressor). Quando
o repressor é sintetizado ele é inativo.
Entretanto, ele pode ser
ativado ao se complexar com o co-repressor (ou seja, o triptófano).
Operador
O complexo repressor/co-repressor ativo age através da ligação a uma região
específica do DNA chamada de operador, que está adjascente aos genes
estruturais que estão sendo regulados. Os genes estruturais juntamente com a
região do operador e o promotor são chamados em conjunto de OPERON. Assim,
na presença do co-repressor o repressor é ativo e se liga ao operador,
resultando na repressão da transcrição dos genes estruturais. Ao contrário,
na ausência do co-repressor o repressor é inativo e não se liga ao operador,
resultando na transcrição dos genes estruturais. Este tipo de controle é
chamado de CONTROLE NEGATIVO, visto que a função do produto do gene
regulatório (repressor) é de desligar a transcrição dos genes estruturais.
Co-repressor
A transcrição dos genes do triptófano é influenciada pela presença ou
ausência de um co-repressor (triptófano) (Figura 7).
|
e.g.
|
+
co-repressor |
Ausência de
expressão |
|
|
-
co-repressor |
expressão |
|
 Figura 8 Mecanismo de atenuação
Figura 8 Mecanismo de atenuação
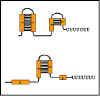 Figura 9 Formação de pareamentos e alças (grampos)
Figura 9 Formação de pareamentos e alças (grampos) |
Atenuação
Em muitos
operons repressíveis, a transcrição que se inicia no promotor pode
terminar prematuramente em uma região lider que precede o primeiro gene
estrutural. (i.e. a polimerase termina a transcrição antes de
chegar ao primeiro gene do operon). Esse fenômeno é chamado de
ATENUAÇÃO; o término prematuro da transcrição. Embora a atenuação seja
vista em vários operons, o mecanismo é melhor entendido naqueles operons
repressíveis envolvidos na biossíntese de aminoácidos. Nesses casos a
atenuação é regulada pela disbonibilidade de um RNAt aminoacilado
cognato.
Mecanismo
(Ver Figura 8)
Quando a transcrição é iniciada no promotor, ela começa na verdade antes
do primeiro gene estrutural e um transcrito lider é feito. Esta região
líder contém um sinal de início e um sinal de parada da síntese
protêica. Uma vez que a bactéria não tem uma membrana nuclear, a
transcrição e tradução podem ocorrer simultaneamente. Assim, um pequeno
peptídio pode ser feito enquanto a RNA polimerase está transcrevendo a
região lider. O peptídio-teste contém alguns resíduos de triptófano no
meio dele. Assim, se tem uma quantidade suficiente de triptofanil-t-RNA
para traduzir o peptídio-teste, o peptídio inteiro será feito e o
ribossoma vai chegar até o sinal de parada. Se, por outro lado, não tem
bastante triptofanil-t-RNA para traduzir o peotídio, o ribossoma vai
parar nos dois codons do triptófano antes de chegar ao sinal de parada.
A sequência no
RNAm lider contém quatro regiões, as quais têm sequências complementares
(Figura 9). Assim, estruturas diferentes secundárias “pareadas e em
alça” podem ser formadas. Região 1 pode formar somente pareamento com a
região 2; região 2 pode formar pareamento ou com a região 1 ou com a
região 3; região 3 pode formar pareamento com a região 2 ou 4; e região
4 pode formar pareamento somente com a região 3. Assim, três estruturas
pareamento/alça podem ser formadas no RNA.
região 1:região
2
região 2: região
3
região 3: região 4
Uma das
estruturas possíveis (região 3 pareando com a região 4) gera um sinal
para a RNA polimerase terminar a transcrição (i.e. para atenuar a
transcrição). Entretanto, a formação de uma estrutura em pareamento e
alça pode eliminar a formação das outras. Se a região 2 forma pareamento
com a região 1 ela não está disponível para parear com a região 3. Da
mesma forma, se a região 3 forma pareamento com a região 2 ela não está
disponível para parear com a região 4.
A habilidade dos
ribossomas de traduzir o peptídio-teste irá afetar a formação das várias
estruturas em pareamento e alça (Figura 10). Se o ribossoma atinge o
sinal de parada da tradução ele vai estar cobrindo a região 2 e assim a
região 2 não vai estar disponível para formar pareamento com outras
regiões. Isso permite a geração do sinal de término da transcrição
porque a região 3 vai estar disponível para parear com a região 4.
Assim, quando tiver bastante triptofanil-t-RNA para traduzir o
peptídio-teste a atenuação vai ocorrer e os genes estruturais não serão
transcritos. Ao contrário, quando tiver quantidadde de triptofanil-t-RNA
insuficiente para traduzir o peptídio-teste não vai acontecer atenuação.
Isso é porque o ribossomo vai parar nos dois codons de triptofano na
região 1, permitindo portanto a região 2 parear com a região 3,
impedindo a formação do sinal de atenuação (i.e. região 3 pareia
com região 4). Assim, os genes estruturais vão ser transcritos.
|
 Figura 10 Mecanismo de atenuação
Figura 10 Mecanismo de atenuação |
| |
REGULAÇÃO DA ATIVIDADE ENZIMÁTICA
Bactéria também tem
maneiras de regular as atividades de suas enzimas.
Inibição por
feedback (inibição por retroalimentação)
A atividade de enzimas bacterianas é frequentemente sujeita a inibição por
feedback. Normalmente é o produto final de uma via que é o inibidor e a primeira
enzima da via é a etapa que é regulada.
Modificação
epigenética
As atividades de enzimas bacterianas podem também ser reguladas por modificações
covalentes de enzimas. Tais modificações são chamadas de MODIFICAÇÕES
EPIGENÉTICAS.
Ex. Adenilação da
glutamina-sintetase
Fosforilação da
glicogênio-sintetase
Normalmente essas modificações são reversíveis, de forma que as atividades das
enzimas podem ser ligadas e desligadas.
|
|
|
 Retorne à Seção de Bacteriologia de Microbiologia e
Imunologia On-line
Retorne à Seção de Bacteriologia de Microbiologia e
Imunologia On-line
Esta página foi traduzida do original em inglês por Myres MTR Hopkins, PhD
em Ciências (Genética – Universidade de São Paulo) e é mantida por Richard
Hunt
Por favor, relate quaisquer problemas para
mmtr5@hotmail.com
|

 Figura
3 Repressão catabólica
Figura
3 Repressão catabólica
 Figura 6
Figura 6  Figura 8 Mecanismo de atenuação
Figura 8 Mecanismo de atenuação





