|
|
 |
||||
|
Dr Richard Hunt |
BACTERIOLOGÍA | INMUNOLOGÍA | MICOLOGÍA | PARASITOLOGÍA | VIROLOGÍA |
|
|||||
|
VIDEOCONFERENCIA |
|||||

|
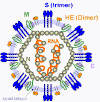
Los coronavirus, que tienen un diámetro aproximado de 100nm, son los virus más grandes de ARN de sentido positivo (de hecho tienen el genoma más grande de todos los virus de ARN). Infectan tanto humanos como animales en los que causan enfermedades respiratorias y entéricas. Los coronavirus, junto a los torovirus y los arterivirus, pertenecen al grupo de los nidovirales, que producen un grupo anidado de ARNm con terminales 3’ en común (vea más adelante). Los coronavirus y los torovirus (que juntos componen a los Coronaviridae) tienen nucleocápsides helicoidales mientras que los arterivirus tienen nucleocápsides icosaédricas. Los coronavirus tienen una envoltura derivada de las membranas intracelulares y no de la membrana plasmática. En las micrografías electrónicas se ven con espinas saliendo de sus superficies (productos de una gran glicoproteína), y que da pie a su nombre (corona) (Figura 1 y 2). En este capítulo sólo se discutirán los Coronavirus puesto que son particularmente importantes en enfermedades respiratorias humanas, causando un tercio de los “resfriados comunes” y causando la nueva entidad conocida como síndrome respiratorio agudo severo (SARS).
|
||||
 Figura 2 Torovirus © Universidad de Queen's en Belfast
|
Proteínas Proteína S (espina) (150k) Esta es una glicoproteína transmembranal con tres dominios (figura 3): el dominio largo externo (con dos sub dominios), la secuencia transmembrana y el pequeño dominio interno. El dominio externo (N-terminal) se pliega para adoptar una forma globular y formar las estructuras de espinas en las micrografías electrónicas. Esta región da al virus sus propiedades antigénicas y contiene el sitio de unión para el receptor de superficie celular. La parte interna del dominio externo está probablemente enrollada como un espiral y contiene repeticiones en serie de siete. Hay una molécula acil-grasa que ayuda a estabilizar la proteína en la bicapa lipídica. La parte interna del dominio externo forma una estructura con forma de talo que se asocia con otras proteínas S para formar un trímero. En algunos coronavirus, el dominio externo es clivado pero las dos partes de la glicoproteína permanecen unida mediante interacciones iónicas (de un modo similar a las gp120 y gp41 del VIH). La parte interna de la proteína S, la cual puede exponerse con una unión a la célula huésped, es responsable de la fusión de membranas. La proteína S tiene una región similar a los receptores Fc-gamma de las inmunoglobulinas permitiendo que el virus se cubra con estas proteínas protegiéndose de ataques inmunes (los herpes virus tienen una estrategia similar). La proteína S puede unirse a ácido siálico (ácido 9-O-acetil neuramínico) en la superficie de la célula huésped lo que proporciona al virus capacidad de hemoaglutinación. Los anticuerpos contra la proteína S son neutralizantes. Proteína HE (65kD) Pocos coronavirus tienen una proteína hemaglutinina-esterasa. Esto también forma espinas (que son más cortas que las espinas S) en la superficie del virus. Es un dímero y no parece ser esencial para la replicación en aquellos tipos que la poseen. Esta proteína también une ácido siálico. La actividad esterasa de la proteína HE puede separar el ácido siálico de la cadena sacarosa, lo cual puede ayudar al virus a escapar de la célula en la que se replicó. Los anticuerpos contra la proteína HE también pueden neutralizar el virus. Proteína M (membrana) Esta es otra glicoproteína que atraviesa la membrana pero gran parte de ‘esta es interna y sólo una pequeña porción con un dominio N-terminal es externa. La proteína M atraviesa la membrana tres veces. Esta proteína ayuda en la fijación de la nucleocápside a la membrana de estructuras internas tales como el cuerpo de Golgi y no se encuentra en la membrana plasmática de la célula (a diferencia de otras glicoproteínas). Proteína E (envoltura) (9-12kD) Esta es una proteína pequeña también de la membrana viral. En la célula infectada se encuentra cerca del núcleo y en la superficie celular. Proteína N (nucleocápside) (60kD) La proteína de nucleocápside se une al ARN genómico mediante secuencias iniciadoras y a la proteína M de la superficie interna de la membrana viral. La proteína N está fosforilada. A diferencia de muchos virus de ARN, los coronavirus no incorporan la ARN polimerasa a la particular vírica; en vez de ello la polimerasa se sintetiza luego de la infección utilizando el ARN genómico de sentido positive como un ARNm. Esto es posible gracias al gen pol en el terminal 5’ del genoma.
|
||||
|
Fijación del virus a la célula huésped
Como se
mencionó antes, la principal proteína de fijación es la proteína S y
esta se une al ácido siálico. La proteína HE también se une al ácido
siálico. Pero, el ácido siálico se encuentra en las superficies de las
células y los coronavirus tienen un tropismo tisular restringido y por
tanto la fijación se hace más complicada. Más aún, algunos coronavirus
no se unen del todo al ácido siálico. La proteína S puede unirse a otros
receptores más específicos. En el caso del virus de la hepatitis de
murinos, el receptor parece ser miembro de la superfamilia de
inmunoglobulinas y cualquier anticuerpo contra esta proteína impedirá la
fijación del virus. Otros coronavirus incluyendo el coronavirus
respiratorio humano usan una metaloproteinasa de membrana
(aminopeptidasa N) como receptor. |
|||||
|
Penetración Fusión de la membrana viral con la membrana celular, un prerrequisito para la replicación viral en el citoplasma, puede ocurrir en la membrana plasmática o en endosomas acídicos.
|
|||||
 Figura 4. Coronavirus dentro de una vacuola de membrana y cisternas del retículo endoplásmico rugoso. Esta delgada sección de TEM de células V6 infectadas, muestra partículas de coronavirus, sospechosos de ser causales del síndrome respiratorio agudo severo (SARS). CDC/C.S. Goldsmith/T.G. Ksiazek/ S.R. Zaki |
Ensamblaje viral Hay una secuencia de 61 nucleótidos cerca del terminal 3’ del genoma que solo se encuentra en el ARN genómico de sentido positivo. Esta interactúa con la proteína N para formar la nucleocápside. La nucleocápside interactúa con la proteína M que es expuesta en las superficies citoplasmáticas de las membranas intracelulares (retículo endoplásmico, cuerpo de Golgi y especialmente un compartimiento entre el RE y el cuerpo de Golgi, quizás la red cis-Golgi). La proteína M no se encuentra en la superficie citoplásmica de la membrana plasmática. Parece que la proteína N debe de estar asociada con el ARN para que esto suceda. Además, la proteína E es necesaria para la yemación de la nucleocápside a la membrana en donde puede alterar la curvatura de la misma durante el proceso de yemación. Es probable la proteína E la que se une con la M. la proteína E se encuentra en el virus maduro, pero sólo en pequeñas cantidades de forma que quizás sólo sea una proteína de andamiaje para la iniciación del ensamblaje y de la que se puede disponer después. Las proteínas S y HE también interactúan en el plano de la bicapa lipídica del compartimiento de yemación con la proteína M. Los complejos S-M y HE-M se asocian luego y las cadenas sacarosas son procesadas a medida que el virus para por el cuerpo de Golgi. El virus madura morfológicamente en el cuerpo de Golgi y se acumula en vesículas de membrana en el citoplasma para luego fundirse con la membrana plasmática (figura 4).
|
||||
|
Genoma Los coronavirus tienen un genoma de cadena sencilla muy grande (para ser virus de ARN), de hecho son los virus más grandes de ARN. El genoma es de sentido positivo (esto es, en el mismo sentido que el ARNm) y no es segmentado (c.f. los ortomixovirus). El ARN genómico está chapado y poliadenilado y tiene entre 27 y 32kB. El gran tamaño del genoma, añadido con la carencia de lecturas de corrección en la ARN polimerasa, lo que hacen que los coronavirus tengan un alta tasa de mutación. Varios coronavirus han sido secuenciados, incluyendo el virus del SARS. El orden de los genes es siempre el mismo. En el terminal 5’ está la polimerasa (pol) y se sigue de cuatro proteínas estructurales de los coronavirus:
Algunos coronavirus también tienen un gen entre el gen pol y el gen S que puede haber sido adoptado de un paramixovirus, el gen de la hemaglutinina-esterasa (HE). También hay marcos de lectura abiertos adicionales (ORFs) que no son muy conservados entre los diferentes coronavirus. Estos genes probablemente codifican para proteínas pero su función todavía es desconocida. Además de los genes codificadores de proteínas, todos los coronavirus tienen secuencias de 7 bases llamadas intergenicas que están en el terminal 5’ de cada gen. Si la secuencia intergenica se altera (muta), el ARNm sub-genómico que inicia en este punto no se sintetiza.
|
|||||
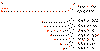 Figura 5. Los ARN mensajeros de los corona virus. Se forma un grupo anidado de ARN con terminales 3’ comunes. Los ARNm para la polimerasa (pol) tienen la misma longitud que el ARN genómico. El resto es truncado en el terminal 5’ aunque todos tienen una secuencia iniciadora común |
Replicación Gran parte del genoma de ARN que las células infectadas está destinado para ser empacado en partículas virales. Estas moléculas son presuntamente hechas por síntesis continua (contrario a los ARNm), aunque hay evidencia de producción por separado de la secuencia iniciadora igual que en la síntesis de ARNm (vea debajo). ARN Mensajero Todos los coronavirus sintetizan un grupo anidado de ARNm que tienen un terminal 3’ en común pero carecen del terminal 5’ (a excepción del ARNm que codifica para el gen de la proteína 1, la polimerasa) (Figura 5). Como el ARN genómico, estos ARNm sub-genómicos son chapados y poliadenilados. Sólo una proteína es traducida de cada ARNm sub-genómico, y esta es la proteína codificada en el marco de lectura abierto (ORF) 5’, no obstante todas estas moléculas, con excepción de la más pequeña, tienen más de una secuencia codificadora de proteína. Cada ARNm también tiene una secuencia iniciadora común de más o menos 70 bases en el terminal 5’. Esto también se encuentra en el terminal 5’ del ARN genómico pero no se encuentra en ninguna otra parte de la molécula, aunque las secuencias intergenicas son similares a la región iniciadora. Cuando el ARN genómico entra al citoplasma, es copiado a una hebra negativa complementaria. Esto luego es copiado de vuelta a una hebra genómica positiva y a ARNm sub-genómicos. Las células también contienen hebras de ARN sub-genómicas negativas (anti-sentido) que siempre están en complejos de cadena doble con hebras de sentido. No se conoce cómo se sintetizan los ARNm sub-genómicos son su secuencia iniciadora común pero se han sugerido diversas posibilidades. Por ejemplo, la secuencia iniciadora de la hebra negativa puede ser copiada a una hebra positiva de cerca de 70 bases. La secuencia iniciadora podría luego disociarse de la hebra genómica y reconocer alguna de las secuencias intergénicas en la plantilla. Aquí, podría imprimar la síntesis de hebras positivas tan largas como el ARN genómico. Una observación que apoya la teoría de este modelo discontinuo de síntesis de ARNm es que si una secuencia intergenica se inserta artificialmente al genoma se forma un ARNm nuevo que inicie en este punto e incluya la secuencia iniciadora.
|
||||
|
La polimerasa (replicasa) El primer gen en el terminal 5’ del genoma es aquel que codifica para la replicasa o ARN polimerasa. Es responsable de más de la mitad del genoma (dado que tiene una longitud de 20kB). La secuenciación muestra que este gen de hecho contiene dos secuencias codificadoras de proteínas que se hallan en diferentes marcos de lectura que se solapan. No obstante, las dos secuencias dan pie a una proteína, llamada poliproteína, mediante disminución ribosómica cuando el ribosoma se acerca al inicio de la secunda secuencia. A medida que se sintetiza la gran poliproteína es separada por proteasas parte de la proteína naciente. Una de las proteínas liberadas es la ARN polimerasa. Mutación y recombinación Los coronavirus tienen genomas grandes de ARN que se replican por replicasa/polimerasa codificadas por el virus. Las ARN polimerasas no tienen capacidad de corrección de lectura y típicamente tiene una tasa de error de casi 1 en 10,000 nucleótidos. Puesto que el genoma de un coronavirus promedio es de más o menos 30kB, esto significa que habrá varias mutaciones en cada progenie del virus. También hay muchas mutaciones por supresión formadas en los coronavirus. Hay una alta frecuencia de recombinación en los coronavirus lo cual no es típico de virus no segmentados de ARN. Esto puede deberse al modo discontinuo de la replicación de ARN en la que la secuencia iniciadora se sintetiza y luego el iniciador/polimerasa “saltan” a otra hebra. Esta alta tasa de recombinación resulta en la rápida evolución del virus y en la formación de nuevas cepas.
|
|||||
|
Resfriados y otros aspectos de las enfermedades por coronavirus Patogénesis Los coronavirus causan enfermedades respiratorias y entéricas en una variedad de animales. En humanos, el principal sitio de replicación viral son las células epiteliales del tracto respiratorio y casi un tercio de los resfriados son causados por coronavirus. Los síntomas son similares a los del resfriado por rinovirus (rinorrea, dolor de garganta, cefalea, fiebre, escalofríos, etc.) con un periodo de incubación de más o menos 3 días. La diseminación viral es limitada por la respuesta inmune de muchos pacientes pero esta inmunidad es de corto plazo. Los síntomas pueden durar por hasta una semana dependiendo del paciente. Con frecuencia no hay síntomas aparentes y el paciente como quiera descama el virus infeccioso. A diferencia de los rinovirus, que no tienen envoltura, los coronavirus son un tanto inestables. La transmisión es por transferencia de secreciones nasales por ejemplo al estornudar. Los virus que infectan las células epiteliales del tracto entérico causan diarrea. Aunque esto puede ocurrir en humanos recién nacido es mas común en animales jóvenes y en estos puede ser fatal. Aunque las infecciones por coronavirus son generalmente locales, pueden diseminarse. En los humanos, estos virus han sido implicados en las infecciones de oído medio, en algunas neumonías en pacientes inmuno-suprimidos y en miocarditis, pero, como ya se mencionó, es mucho más severa en animales (e.g. peritonitis felina infecciosa). Interesantemente, los coronavirus, que en el laboratorio pueden infectar células neurológicas, pueden causar una enfermedad en los roedores que es muy similar a la esclerosis múltiple, lo que ha llevado a la sugerencia de que están implicados con dicha enfermedad en humanos; la demielinización, una característica de la esclerosis múltiple en el modelo de los roedores, está ligada con la proteína S y se ha sugerido que la enfermedad resulta de similitudes moleculares en las que la respuesta inmune que habría de dirigirse a la proteína S se dirige a la mielina. Sin embargo, aunque el virus puede ser detectado en el cerebro de los pacientes, el eslabón con la esclerosis múltiple todavía queda por comprobarse.
|
|||||
|
Epidemiología Muchas personas albergan anticuerpos anti-coronavirus pero las reinfecciones son comunes lo cual es indicativo de que hay muchos serotipos circulantes del virus en la población humana. No parecen haber reservorios animales para aquellos virus que infectan humanos. Como sucede con la mayoría de las infecciones respiratorias, los resfriados causados por coronavirus son más comunes durante el invierno porque hay más contactos cercanos. Los brotes más grandes se dan cada cierta cantidad de años con un ciclicidad que depende del tipo de virus implicado. Diagnóstico La mayoría de las infecciones por coronavirus pasan sin diagnosticarse y la enfermedad es auto limitante. El diagnóstico puede realizarse usado microscopio inmunoelectrónico y serología. No hay una rutina de antivirales para las infecciones por coronavirus pero hay medicamentos sin prescripción que alivian los síntomas.
|
|||||
|
FUENTES
EN LA RED Inst. de Patología de las Fuerzas Armadas de los EU: Reportes de casos de SARS |
|||||
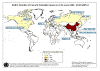 Figura 6A. Mapa de probables casos de SARS. Junio 02, 2003 OMS 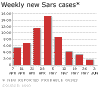 Figura 6B. Casos nuevos semanas de SARS. © OMS/BBC
|
Síndrome respiratorio agudo severo (SRAS o SARS) A finales del 2002, se observó un nuevo síndrome en el Sur de China (en la provincia de Guangdong). Se denominó síndrome respiratorio agudo severo (SARS). Esta enfermedad, que ya ha sido reportada en Asia, América del Norte y Europa (figura 6A), se caracteriza por fiebre de más de 38 grados (100.4 grados Fahrenheit) acompañada de cefalea, malestar general, y dolores. De hecho, los síntomas respiratorios iniciales son leves durante unos días (o una semana), el paciente desarrolla una tos no productiva y la respiración se le hace dificultosa (disnea). El distress respiratorio lleva a la muerte en un 3-30% de los casos. Los exámenes de laboratorio evidencian una reducción en el número de linfocitos y un aumento en la actividad de las aminotransferasas lo cual indica daño hepático. El pico inicial del brote de SARS fue en Abril 2003 y había disminuido para Junio. Pero para ese tiempo, había casi 8,000 casos a nivel mundial y 775 muertes. El virus fue cultivado en células Vero E6 de monos en cultivos titulares y un nuevo coronavirus (SARS-coV) fue asociado con la enfermedad. Tiene un genoma de 29,727 bases y 11 marcos de lectura abiertos (ORFs). La secuencia, aunque similar a la de otros coronavirus, es lo suficiente diferente como para hacer de este miembro un nuevo grupo de coronavirus. La organización del genoma es muy parecida a la de otros coronavirus (5’ replicasa (rep), espina (S), envoltura (E), membrana (M), nucleocápside (N)-3′ y pequeñas regiones no traducidas en ambos terminales). El gen de la replicasa ocupa los dos tercios 5’ del genoma y, como otros coronavirus, tiene dos marcos de lectura abiertos que se solapan. También codifica para una proteasa en la poliproteína pol. Hay nueve posibles marcos de lectura abiertos que no se hallan en otros coronavirus y que codifican proteínas que son únicas del virus del SARS. Usando exámenes de laboratorio, el coronavirus del SARS se ha asociado a sus casos a nivel mundial. Diagnóstico El Centro de Control y Prevención de Enfermedades de los EUA recomiendo radiografías de tórax (figure 8), oximetría de pulso, cultivos sanguíneos, tinción Gram del esputo y cultivo, y evaluación de patógenos respiratorios virales, en especial influenza A y B y el virus sincitial respiratorio. Un examen para el espécimen de Legionella y del antígeno pneumocóccico urinario también debe ser considerados. Las personas en las que se sospecha SARS deben de ser aisladas y puestas en cuarentena. Tratamiento No se ha acordado en ningún tratamiento para el SARS que no sea el manejo sintomático. Existen drogas en desarrollo y estudio y de interés particular son las drogas que bloquean la función de la proteasa puesto que esto es crucial para el virus. No existe vacuna para el virus del SARS o para ningún otro coronavirus. Los programas de inmunización veterinaria han tenido cierto éxito con respecto de ciertos Coronavirus económicamente importantes. Un gran problema con la vacuna es el cambio antigénico y los resultados impredecibles.
|
||||
|
LIBRO WEBCAST Casos de Estudio del SARS en Salud Pública |
|||||
|
|
|||||
|
|||||