|
x |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGÍA |
INMUNOLOGÍA
|
MICOLOGÍA |
PARASITOLOGÍA |
VIROLOGÍA |
|
EN INGLÉS
|
VIROLOGÍA – CAPÍTULO 4
ESTRATEGIAS DE REPLICACIÓN DE LOS VIRUS DE ARNDr Margaret Hunt
University of South Carolina School of Medicine
Columbia
SC
USA
Traducido por :
Sarah M. Castillo -
Jorge
Medico Pasante
Clinica Corominas
Santiago
Rep. Dominicana
|
|
BUSCAR |
|
|
|
|
|
|
|
OBJETIVOS
Análisis descriptivo de las estrategias de replicación empleadas por los
virus de ARN
Identificación de los prototipos de virus asociados a diversos esquemas
de replicación de los virus de ARN
 Estructura del Polio tipo 1 Mahoney.
Datos radiográficos de Hogle et al.(Univ. De Harvard), PDB entry 2PLV,
rendered with GRASP (A.Nicholls, Univ. de Columbia.). Cortesía del Dr.
Sgro
y el
Institute for
Molecular Virology, Univ. de Wisconsin (usado con
autorización)
Estructura del Polio tipo 1 Mahoney.
Datos radiográficos de Hogle et al.(Univ. De Harvard), PDB entry 2PLV,
rendered with GRASP (A.Nicholls, Univ. de Columbia.). Cortesía del Dr.
Sgro
y el
Institute for
Molecular Virology, Univ. de Wisconsin (usado con
autorización) |
REPLICACIÓN DE LOS VIRUS DE ARN - GENERALIDADES
ESTRATEGIAS
VIRUS DE ARN QUE NO POSEEN UNA
FASE DE ADN
Los virus que se replican vía intermediarios de ARN necesitan una ARN –
polimerasa dependiente de ARN para replicar su ARN, pero las células
animales aparentemente no tienen esa enzima. Por tanto, este tipo de virus
de ARN necesita codificar su ARN – polimerasa dependiente de ARN.
Ninguna proteína viral puede ser sintetizada hasta que el ARN mensajero del
virus esté disponible. Por tanto, la naturaleza del ARN en el virión afecta
la estrategia del virus:
Virus
de ARN de cadena positiva
En
estos virus, el ARN del virión (genoma) tiene el mismo sentido que un ARNm y
por tanto funciona como tal. Este ARNm puede ser traducido inmediatamente
luego de la infección de la célula huésped
Ejemplos:
Virus de ARN de cadena negativa
El ARN
del virión tiene sentido negativo (complementario al ARNm) y debe de ser,
por tanto, copiado en un ARNm de sentido positivo complementario antes de
que las proteínas puedan ser sintetizadas. Entonces, aparte de necesitar
codificar para una ARN – polimerasa dependiente de ARN, estos virus
necesitan también empacar la misma en el virión para tener la
posibilidad de sintetizar ARNm cuando infectan una célula.
Ejemplos:
Virus de ARN de cadena doble
El ARN
del virión (genómico) es de cadena doble y por eso no puede funcionar como
ARNm; estos virus necesitan también empacar una ARN – polimerasa para
sintetizar su ARNm luego de producir infecciones en células huésped.
Ejemplo:
VIRUS DE ARN QUE COPIAN SU ARN EN ADN
Estos
son los retrovirus. En este caso, el ARN del virión, aunque tiene sentido
positive, no funciona como ARNm inmediatamente luego de la infección puesto
que no es liberado de su cápside al citoplasma. Al contrario, sirve de
plantilla para la transcriptasa inversa y es copiado en ADN. La
transcriptasa inversa no está disponible en la célula, y por eso, estos
virus necesitan codificarla y empacarla en los viriones.
|
VIRUS DE ARN QUE NO TIENEN FASE
DE ADN |
|
Genoma |
ARN – polimerasa dependiente de ARN (=transcriptasa) en virión
|
Contagiosidad del ARN |
Evento inicial en la célula |
|
ARN de cadena positive |
No |
Infeccioso |
Traducción |
|
ARN de cadena negativo |
Si |
No infeccioso |
Transcripción |
|
ARN de cadena doble |
Si |
No infeccioso |
Transcripción |
|
RETROVIRUS |
|
Genoma |
ARN – polimerasa ARN dependiente (=transcriptasa) en virión |
Contagiosidad del ARN |
Evento inicial en la célula |
|
ARN de sentido positivo |
Si |
No infeccioso |
Transcriptasa inversa |
EL PROBLEMA DE TRADUCCIÓN
La
maquinaria de síntesis de proteínas de traducción de las células huésped
eucarióticas generalmente utiliza ARNm
monocistrónicos y
por eso, hay un problema al hacer más de un tipo de proteína de un único ARNm.
Los virus
de ARN tienen diversas soluciones a este problema:
i. El virus
sintetizan muchos ARNm monocistrónicos
ii. El
virus hace transcripciones primarias que son procesadas por la maquinaria de
empalme del huésped para tener más de un ARN monocistrónico
iii. El
ARNm viral actúa como una transcripción monocistrónica. Se sintetiza una
poliproteína que luego es separada – así, un producto inicial de la traducción
es procesado para que surjan múltiples proteínas. Esto sucede, por ejemplo, en
los picornavirus.
iv. El ARNm viral
tiene funciones especiales que activan ribosomas para que se unan internamente y
no al terminal 5’.
TAMAÑO DEL GENOMA DE LOS VIRUS DE ARN
Los virus
de ARN tienden a tener genomas relativamente pequeños (aun cuando el tamaño del
virión no sea necesariamente pequeño). Esto probablemente es porque la ausencia
de los mecanismos de corrección de errores en el ARN limita el tamaño de los
genomas de ARN.
Como
consecuencia del pequeño tamaño, los virus de ARN tienden a codificar tan solo
unas cuantas proteínas. Entre las cuales se incluye una polimerasa que puede
copiar ARN en un ácido nucleico complementario (sea ARN, o en el caso de los
retrovirus, ADN) y una proteína de adhesión viral
|
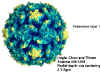 Figura
1 Polio virus
© 1999
Dr. J-Y Sgro,
Centro de Biotecnología de la Universidad de Wisconsin, Fuentes de Gráficas
Moleculares. Usado con autorización.
Figura
1 Polio virus
© 1999
Dr. J-Y Sgro,
Centro de Biotecnología de la Universidad de Wisconsin, Fuentes de Gráficas
Moleculares. Usado con autorización.
 Figura 2 Polio virus x350,000
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
Figura 2 Polio virus x350,000
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
|
VIRUS DE ARN DE CADENA POSITIVA
Ejemplos:
-
picornavirus
-
togavirus
-
flavivirus
PICORNAVIRUS
PROPIEDADES
Estos son
virus pequeños (28nm), desnudos e icosaédricos (figura 1) (pico= muy pequeño).
El ARN es de cadena sencilla, de sentido positive y poliadenilado. Funciona como
una ARNm inmediatamente luego de la infección.
Miembro prototipo: poliovirus (figuras 1 y 2)
FIJACIÓN Y
PENETRACIÓN
Una
proteína viral reconoce un receptor en la membrana plasmática de la célula
huésped (esto es importante en el tropismo del virus).
Parece ser que la unión al receptor altera la estructura de la cápside de alguna
manera, se forma un canal a través de la membrana plasmática y el ARN es
liberado dentro del citoplasma. Ahora hay un ARNm disponible para la traducción.
SÍNTESIS DE
LAS PROTEÍNAS VIRALES
El ARN del
virión del poliovirus funciona como una ARNm pero no tiene una estructura
metilada tipo chapa típica de los ARNm eucarióticos – posee un “ regazo de
aterrizaje para ribosomas” (conocida como el sitio de entrada interno de los
ribosomas o IRES por sus siglas en ingles), el cual permite a los ribosomas el
unirse sin tener que reconocer la chapa metilada en 5’ (figura 3).
Los picornavirus a menudo interfieren con el reconocimiento de la chapa metilada
de la célula huésped. Muchas traducciones de la célula huésped dependen de esta
chapa, por tanto inhiben muchas de las síntesis proteicas del huésped (no así la
síntesis de proteínas víricas) – ésta es una forma en la que estos virus
modifican a la célula huésped para su conveniencia.
|
 Figura 3
Figura 3
|
El ARNm es traducido en una cadena polipeptídica sencilla (poliproteína), la
cual es separada. Las divisiones ocurren antes de que la traducción se complete
(i.e. en la cadena naciente (creciente)) y son transportadas afuera por
proteasas codificadas por el virus (figura 4). Algunas de estas proteasas pueden
trabajar aún estando alejadas de la poliproteína.
|
 Figura 4 Adaptada de Schaechter et al., Mechanisms of Microbial Disease, 2a
Ed.
Figura 4 Adaptada de Schaechter et al., Mechanisms of Microbial Disease, 2a
Ed. |
Los
productos de la separación de la poliproteína incluyen:
Una ARN
polimerasa (replicasa)
Componentes estructurales del virión
Proteasas
|
 Figura 5
Figura 5 |
REPLICACIÓN
DE ARN
Ahora se
tienen proteínas virales recién sintetizadas para ayudar en la replicación.
1. ARN
polimerasa viral copia el ARN genómico de sentido positivo en un ARN
complementario de sentido negativo:
Este proceso requiere de
VPg -
proteína viral g (o precursores que contengan VPg)
ARN polimerasa viral (replicasa)
Ciertas proteínas de la célula huésped
La VPg
puede actuar como un iniciador para la síntesis de ARN, esto explicaría porqué
se encuentra en la Terminal 5’ de todas las moléculas de ARN recién sintetizadas.
2. Las
nuevas cadenas de sentido negativo sirven como plantilla para nuevas cadenas de
sentido positivo (figura 5). Otra vez, se necesitan la ARN polimerasa del polio
y la VPg. La VPg esta unida al Terminal 5’ de las nuevas cadenas de sentido
positivo (y, una vez más, probablemente funcione como un iniciador).
La nueva cadena
de sentido positivo tiene 3 destinos alternativos:
i. Puede servir como plantilla para crear más cadenas de sentido negativo
ii. Puede ser empacada en los viriones progenie
iii. Puede ser traducida a poliproteína (En este caso la VPg es, generalmente,
removida previo a la traducción)
ENSAMBLAJE
Cuando se
ha acumulado suficiente cantidad de ARN de sentido positivo y de proteínas del
virión, el ensamblaje comienza. Las partículas se ensamblan con ARN-VPg adentro
y con 3 proteínas en la cápside [VP0,1 y 3]. Luego VP0 es separada en VP2 y VP4
a medida que el virión adquiere madurez y se hace infeccioso. Los viriones se
liberan luego de la lisis celular. Se forman cápsides en exceso y entonces
algunos cuerpos de inclusión pueden ser vistos en el citoplasma.
NOTA: EL
CICLO VITAL COMPLETO OCURRE EN EL CITOPLASMA
NO HAY
DIVISIÓN DE LA EXPRESIÓN DE LOS GENES EN FASES TEMPRANA NI TARDÍA
|
 Figura 6 Rabdovirus en una célula epitelial de un pez
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
Figura 6 Rabdovirus en una célula epitelial de un pez
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
|
VIRUS DE CADENA NEGATIVA NO SEGMENTADOS
Ejemplos:
-
Rabdovirus (figura 6)
-
Paramyxovirus (figura 11)
|
 Figura
7 Estructura de un rabdovirus típico
Figura
7 Estructura de un rabdovirus típico
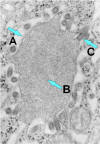 Figura 7b
Figura 7b
Rabdovirus yemando de una inclusión (cuerpo de Negri) hacia el retículo
endoplásmico en una célula del sistema nervioso. A. Cuerpo de Negri. B.
Note la abundante ribonucleoproteína en la inclusión. C. Rabdovirus yemando.
CDC |
RABDOVIRUS
Ejemplo:
Virus de la rabia. El miembro más estudiado es el virus de la estomatitis
vesicular.
El ARN es:
de cadena sencilla
de sentido negativo
codifica 5 proteínas
FIJACIÓN,
PENETRACIÓN Y PÉRDIDA DE LAS ENVOLTURAS
El virus se fija a la superficie celular.
La proteína G (Glicoproteína) es la proteína de fijación (figura 7) la cual se
une a un receptor de la superficie de la célula huésped.
El virus fijado es engullido por endocitosis.
La membrana del virus se funde con la membrana del endosoma (el pH ácido del
endosoma es importante puesto que la proteína G necesita ser expuesta a un pH
ácido para facilitar la fusión).
Como resultado de la fusión de la envoltura viral con la membrana del endosoma,
la nucleocápside es liberada dentro del citoplasma.
TRANSCRIPCIÓN
El término 'transcripción' es utilizado en este contexto para referirse a la
síntesis de ARNm.
La pérdida completa de la nucleocápside no es necesaria para la transcripción –
la ARN polimerasa del virión pueden copiar el ARN del mismo aún cuando este esté
en la nucleocápside. Esto es una ventaja por el hecho de que entonces el ARN
genómico está protegida de las ribonucleasas.
Hay un ARNm monocistrónico para cada una de las cinco proteínas codificadas por
el virus (figura 8). Los ARNm son chapados, metilados, y poliadenilados. Como
esto ocurre en el citoplasma, ya que son virus ARN con cadena en sentido
negativo, las enzimas para la síntesis y modificación de ARNm están empacadas
con el virión.
TRADUCCIÓN
Los ARN
mensajeros son traducidos en los ribosomas de la célula huésped y las cinco
proteínas virales son sintetizadas a un tiempo. No hay distinción de funciones
en tempranas o tardías.
|
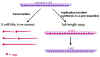 Figura 8
Figura 8 |
REPLICACIÓN
DE ARN
La
replicación del ARN es el proceso mediante el cual nuevas copias de ARN son
sintetizadas (figura 8).
La replicación de ARN se da en el citoplasma y es llevada a cabo por las ARN
polimerasa víricas.
La cadena positiva en su totalidad es envuelta por una proteína de nucleocápside
mientras se va sintetizando (el ARNm no es envuelto con esta proteína, porque
esto interrumpiría con la maquinaria de traducción de la célula huésped).
Las recién
sintetizadas cadenas de sentido positivo son copiadas en su totalidad en cadenas
de sentido negativo, la misma también es envuelta con proteína de nucleocápside
mientras es sintetizada. (Nota: como la ARN polimerasa viral sintetiza ARNm (transcripción) y también ARN completo (replicación), se le conoce también como
transcriptasa o replicasa, estas denominaciones sólo se enfocan en los
diferentes aspectos de la actividad de la polimerasa.)
Las cadenas
negativas recién sintetizadas pueden:
i. Ser
usadas como plantillas para la síntesis de más cadenas enteras de sentido
positivo
ii. Ser usadas como plantillas para la síntesis de más ARNm
iii. Ser empacadas en los viriones
|
 Figura 9
Figura 9
|
ENSAMBLAJE
El virus
consiste en dos “módulos” – la envoltura y la nucleocápside:
Envoltura
Las proteínas transmembranales son sintetizadas en los ribosomas que se
encuentran adyacentes al retículo endoplásmico. Mientras son sintetizadas son
insertadas en retículo endoplásmico en donde son glicosiladas y luego
transportadas al aparato de Golgi en donde se dan modificaciones sustanciales de
las cadenas de carbohidratos. Posteriormente, son transportadas, en vesículas,
hacia la membrana celular apropiada; en el caso del virus de la estomatitis
vesicular, ésta es la membrana plasmática (figura 9).
|
 Figura 10
Figura 10
|
Nucleocápside
La síntesis
de la nucleocápside fue descrita anteriormente. El complejo de ARN polimerasa
vira se asocia con las nucleocápsides cuando se están sintetizando. Las
nucleocápsides yeman a través de áreas modificadas de la membrana, las cuales
contienen proteínas G y M (figura 10). La proteína M (matriz) está involucrada
en el ensamblaje – interactúa con parches de G en la membrana y con
nucleocápsides.
NOTA:
-
TODO EL CICLO DE VIDA
OCURRE EN EL CITOPLASMA
-
LA ARN POLIMERASA Y
LAS ENZIMAS DE MODIFICACIÓN D ARN CON CODIFICADAS POR EL VIRUS MISMO Y ESTÁN
PRESENTES EN EL VIRIÓN
NO HAY
DIVISIÓN EN FASES TEMPRANA/TARDÍA
|
 Figura 11 Paramixovirus ©
Dr. Linda Stannard,
Universidad de Cape Town, Sudáfrica (usado con autorización)
Figura 11 Paramixovirus ©
Dr. Linda Stannard,
Universidad de Cape Town, Sudáfrica (usado con autorización)
|
PARAMIXOVIRUSES
Los
paramixovirus (figura 11) son pleomórficos, esto es: existen múltiples
formas morfológicas en la población viral. Tienen ARN no segmentado de
sentido negativo y una nucleocápside helicoidal (figura 12). Son envueltos,
es decir están rodeados por una envoltura derivada de una célula huésped.
La envoltura contiene dos glicoproteínas codificadas por el virus: La
proteína F y la proteína de fijación
-
La proteína F
tienen implicación en la fusión
-
La proteína de
fijación se une a receptores en la célula huésped
Esta proteína puede tener:
Actividad
de
hemoaglutinación y actividad de
neuraminidasa
(proteína HN) o únicamente poseen actividad de hemoaglutinación (proteína
H) o ninguna de las dos (proteína G).
|
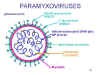 Figura
12 Estructura de un paramixovirus típico
Figura
12 Estructura de un paramixovirus típico |
|
FAMILIA PARAMIXOVIRUS |
|
GÉNERO
|
GLICOPROTEÍNA
|
MEMBROS TÍPICOS
|
|
Paramixovirus
|
HN, F
|
VPIH 1
VPIH 3
|
|
Rubulavirus |
HN, F |
VPIH 2
VPIH 4
virus de la parotiditis |
|
Morbillivirus
|
H, F
|
Virus del sarampión
|
|
Pneumovirus
|
G, F
|
Virus sincitial respiratorio
|
VPIH – Virus de la Parainfluenza humano
Hemoaglutinación
es fácil de detectar en el laboratorio clínico y se usa en el
diagnóstico
Hemoaglutinación
implica
la aglutinación de las células sanguíneas rojas. Se basa en la habilidad del
virus para unirse a receptores en los eritrocitos. Dado que los virus tienen
muchas proteínas de fijación según el virión, pueden adherirse a más de un
eritrocito y por tanto servir como enlace de los mismos en una especie de
red. Incluso un virus inactivo puede hemoaglutinar siempre y cuando sus
proteínas de fijación estén intactas.
Si una
persona posee anticuerpos para las hemoaglutininas virales, estos
anticuerpos se unirán a las proteínas de fijación para prevenir su unión a
los eritrocitos. El suero de esa persona va a inhibir la reacción de
hemoaglutinación de ese virus – pero no así la hemoaglutinación de aquellos
de los cuales no posee anticuerpos. Esta característica puede ser usada para
determinar a qué virus hemoaglutinante una persona ha sido expuesta.
Hemadsorción
Durante una
infección, la proteína de adhesión viral será insertada en la membrana
plasmática de la célula infectada. Si la proteína de adhesión puede unirse a
los eritrocitos, la célula infectada se unirá a eritrocitos porque tendrá la
proteína de adhesión en su superficie – esto se denomina hemadsorción. En el
laboratorio clínico, esto puede activar células infectadas por un virus para
que ser detectadas en fases tempranas de la infección, y puede permitir la
detección de virus que no alteran la célula visiblemente.
|
 Figura 13
Figura 13 |
FIJACIÓN Y
PENETRACIÓN
La proteína
H(N)/G reconoce receptores en la superficie celular.
La proteína
F facilita la fusión de membranas a un pH fisiológico, así que aunque algunos
paramixovirus pueden penetrar por fosas revestidas de la membrana, también
muchas veces pueden penetrar la célula mediante fusión directa con la membrana
plasmática (figura 13).
Gracias a que la
proteína F trabaja a un pH fisiológico, se da la formación de
sincitios
con las infecciones por paramixovirus (véanse las discusiones de las
consecuencias de la fusión a pH fisiológico bajo el acápite de estrategias de
replicación de los virus de ADN – herpesvirus).
|
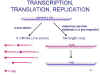 Figura 14
Figura 14 |
TRANSCRIPCIÓN, TRANDUCCIÓN, Y REPLICACIÓN DE
ARN
Los eventos
que ocurren en la célula son similares a los de los rabdovirus (figura 14):
-
La multiplicación
viral ocurre en el citoplasma.
-
La ARN
polimerasa viral utiliza la nucleocápside como plantilla.
-
La ARN polimerasa no
necesita la pérdida completa de la nucleocápside (descapsidación).
-
Los ARNm virales son
transcritos; estos son chapados, metilados y poliadenilados.
-
Como es un
virus de ARN de cadena negativa, las enzimas de modificación del ARN son
empacadas en el virión.
-
Los ARNm virales son
traducidos para sintetizar proteínas virales.
-
No hay división de
las funciones de expresión genética en fases tardía ni temprana.
La
replicación del ARN viral RNA implica la síntesis completa de cadenas de sentido
positivo. Esto es usado como plantilla para cadenas completas en sentido
negativo. Ambas cadenas son envueltas con proteína de nucleocápside mientras son
sintetizadas (figura 14).
Las recién
sintetizadas cadenas de sentido negativo pueden servir a su vez como plantillas
para le replicación, o para la transcripción, o pueden ser empacadas con nuevos
viriones.
|
 Figura 15
Figura 15
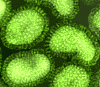 Figura 16 Ortomixovirus (Influenza A)
© Dr. Linda Stannard, Universidad de Cape Town, Sudáfrica
Figura 16 Ortomixovirus (Influenza A)
© Dr. Linda Stannard, Universidad de Cape Town, Sudáfrica
|
ENSAMBLAJE
Ambas
glicoproteínas virales (i.e. la proteína de fijación y la proteína F (de fusión))
son traducidas como proteínas transmembranales y transportadas a la membrana
plasmática de la célula.
La proteína M (matriz) habilita las nucleocápsides para que interactúen con
regiones de la membrana plasmática que tienen las glicoproteínas insertadas.
El virus yema a través de la membrana.
PAPEL DE LA
NEURAMINIDASA
En aquellos
paramixovirus que la poseen, la neuraminidasa facilita su liberación. Es estos
virus, el ácido siálico es, aparentemente, parte importante del receptor. La
neuraminidasa remueve el ácido siálico (ácido neuramínico) de la superficie
celular. Entonces, puesto que el ácido siálico es removido tanto de la
superficie celular como de los viriones progenie, ninguno tendrá receptores
funcionales, y entonces los viriones progenie no se adherirán unos a otros o a
las células de las que yeman (o a otras células infectadas). Estos estarán, por
tanto, en capacidad de difundirse hasta encontrar una célula no infectada.
La
neuraminidasa también puede contribuir con la infección, dado que, si el virus
se une al ácido siálico del mucus, no pudiera unirse a los receptores en la
célula e infectarla. Pero si el ácido siálico en el mucus es destruido
eventualmente, el virus estará libre y podrá alcanzar un receptor en la
superficie celular.
ACTIVACIÓN
DE LA PROTEÍNA F
La proteína
F necesita ser separada antes de que pueda funcionar en la facilitación de la
fusión cuando un virus se une a otra célula (figura 15). Este es un evento
tardío en la maduración.
|
Algunas diferencias entre los rabdovirus y los paramixovirus |
| |
Rhabdovirus |
Paramyxovirus |
|
Forma |
De bala
baciliforme |
ovalada
pleomórfico |
|
Glicoproteínas |
Una (con actividad tanto de fijación como de fusión) |
Dos (una de fijación y una de fusión) |
|
pH de fusión |
acídico |
neutro
fisiológico |
|
|
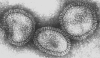 Figura 17 Orthomyxovirus (Influenza A)
© Dr. Linda Stannard, Universidad de Cape Town, Sudáfrica
Figura 17 Orthomyxovirus (Influenza A)
© Dr. Linda Stannard, Universidad de Cape Town, Sudáfrica
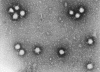 Figura 18 Bunyavirus
De la base de datos del ICTV
Figura 18 Bunyavirus
De la base de datos del ICTV
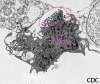 Figura 19b
Vero E6
cultivo tisular célula infectada con un arenavirus. La imagen muestra
partículas víricas extracelulares yemando de la superficie celular.
Magnificación aprox. 12,000 veces.
Figura 19b
Vero E6
cultivo tisular célula infectada con un arenavirus. La imagen muestra
partículas víricas extracelulares yemando de la superficie celular.
Magnificación aprox. 12,000 veces.
Imagen cortesía de Cynthia Goldsmith, MS, Infectious Disease Pathology
Activity, DVRD, NCID, CDC |
VIRUS SEGMENTADOS DE CADENA NEGATIVA
Ejemplos:
ORTOMIXOVIRUSES
Hay
tres grupos de virus influenza: A, B y C. El virus de la influenza A es el
más estudiado y las influenzas A y B son las enfermedades en humanos más
importantes.
Los
virus de la influenza son pleomórficos (esto es, varían en su forma). Tienen
sentido negativo, ARN de cadena sencilla y un genoma de ARN que es
SEGMENTADO. Hay ocho segmentos de ARN en el virus de la influenza A. La
nucleocápside es helicoidal (figura 19). Los viriones contienen ARN
polimerasa empacada dentro de la partícula vírica.
Estos virus son envueltos y tienen dos membranas de glicoproteínas (figura
19):
|
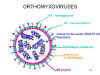 Figura 19 Estructura de un ortomixovirus típico
Figura 19 Estructura de un ortomixovirus típico |
FIJACIÓN Y PENETRACIÓN
El virus se fija a receptores en la superficie celular y es engullido
mediante endocitosis. Al pH ácido del endosoma, HA sufre un cambio
conformacional y ocurre la fusión. Las nucleocápsides son liberadas al
citoplasma.
|
 Figura 20
Figura 20 |
TRANSCRIPCIÓN, TRADUCCIÓN Y REPLICACIÓN
Las
nucleocápsides son transportadas hacia el núcleo. La síntesis del ARNm y la
replicación del ARN viral ocurren en el núcleo. Esto es muy inusual para un
virus de ARN. El virus de la influenza posee un mecanismo poco común para la
adquisición de un Terminal 5’ chapado y metilado para sus ARNm.
Una
endonucleasa viral (la cual es empacada con el virus de la influenza) desprende
del Terminal 5’ del huésped un ARNm chapado y metilado de 13-15 bases y lo
utiliza como un iniciador para la síntesis de ARNm viral (figura 20) – por tanto
todos los ARNm de influenza tienen una corta sección en el terminal 5’ que
deriva de ARN del huésped.
La ARN
polimerasa viral (transcriptasa) extiende el iniciador y copia la plantilla a un
ARNm complementario de sentido positivo y añade un cola poli(A). La
transcripción resulta en 8 transcripciones primarias, una por segmento. Dos de
estos segmentos surgen de transcritos primarios que pueden ser empalmados
alternativamente (puesto que la síntesis del ARN del virus de la influenza
ocurre en el núcleo, tienen acceso a la maquinaria de empalme), cada uno resulta
en dos transcritos alternativos. Por ejemplo, el segmento M resulta en dos ARNm
alternativos. Estos codifican a las proteínas M1 y M2. Por tanto un único
segmento puede codificar a más de una proteína puesto que el virus tiene acceso
a la maquinaria de empalme. Los ARNm son traducidos en el citoplasma. Las
proteínas transmembranales son llevadas a la membrana plasmática mientras que
las proteínas que son necesarias para la replicación del ARN son llevadas al
núcleo.
|
| |
REPLICACIÓN
DEL ARN
La replicación de ARN ocurre en el núcleo usando un enzima codificada por el
virus (esta puede ser igual a la ARN polimerasa de la transcripción de ARNm, o
una versión modificada). Se sintetiza un copia complementaria completa del
virión de ARN – este ARN de sentido positivo es probablemente envuelto en
proteína de nucleocápside mientras se sintetiza. Las recién sintetizadas cadenas
negativas pueden ser usadas como plantillas para replicación, síntesis de ARNm,
o empacamiento.
ENSAMBLAJE
Esto ocurre en la membrana plasmática. Las nucleocápsides son transportadas
fuera del núcleo y las proteínas de envoltura por el aparato de Golgi hacia la
membrana plasmática. La proteína M1 interactúa tanto con la nucleocápside como
con la región modificada de la membrana que contiene las glicoproteínas HA y
NA. Luego el virus yema a través de la membrana de la célula huésped.
Nota:
-
La HA necesita ser separada antes de
que pueda promover la fusión. El requisito de separación afecta cuáles tejidos
pueden producir un virus infeccioso. La proteína separada necesita luego sufrir
un cambio conformacional, usualmente causado por exposición al ambiente del
endosoma acídico cuando infecta una siguiente célula, antes de que pueda ocurrir
fusión.
-
La NA
probablemente ayude al virus liberarse de la célula al remover ácido siálico de
los receptores. La NA puede también ayudar al virus a penetrar mucus y alcanzar
las células epiteliales del tracto respiratorio cuando provocan la destrucción,
en el mismo, de receptores que contienen ácido siálico. La neuraminidasa no
previene que el virus infecte células nuevas puesto que el proceso de
endocitosis supuestamente es más rápido que la remoción de los receptores.
Existen similitudes y diferencias entre las familias Paramixovirus y
Ortomixovirus, integrantes de ambas están envueltos, ambas contienen ARN de
cadena sencilla en sentido negativo, tienen nucleocápsides helicoidales. Sin
embargo, las dos familias son muy diferentes. NO existe relación inmunológica
entre las dos.
|
| |
|
PROPIEDAD
|
PARAMIXOVIRIDAE
|
ORTHOMIXOVIRIDAE
|
|
Genoma
|
No segmentado
|
Segmentado
|
|
Síntesis de RNA
|
Citoplásmico
|
Nuclear
|
|
Necesidad de un iniciador de ARN
|
No
|
Sí
|
|
Hemaglutinina, neuraminidasa
|
Si posee ambas, son parte de una misma proteína (HN)
|
Influenza A y B tienen ambos pero en dos proteínas diferentes (HA y NA)
|
|
Formación de un sincitio
|
Sí
(la proteína F funciona a un pH fisiológico normal)
|
no (HA funciona a un pH ácido)
|
|
 Figura 21 Virión de un reovírus de mamífero
Deerechos reservados 1999
Dr. J-Y Sgro
– Centro de Biotecnología de la Universidad de Wisconsin, Reserva de
Gráficas Moleculares
Figura 21 Virión de un reovírus de mamífero
Deerechos reservados 1999
Dr. J-Y Sgro
– Centro de Biotecnología de la Universidad de Wisconsin, Reserva de
Gráficas Moleculares |
VIRUS DE ARN DE CADENA DOBLE
FAMILIA REOVIRUS
La
familia de los reovirus incluye:
-
A los miembros del género reovirus
-
A los miembros del género rotavirus
-
A los miembros del género orbivirus
-
El Virus de la Fiebre de Garrapata de Colorado
|
 Figura 22 Estructura de un reovirus típico
Adaptado de Joklik et al. Zinsser Microbiology 2Oava. Ed.
Figura 22 Estructura de un reovirus típico
Adaptado de Joklik et al. Zinsser Microbiology 2Oava. Ed. |
Los
reovirus tienen una morfología icosaédrica y una cápside de capas múltiples (cápside
interna y externa) (figura 22)
El ARN es de cadena doble. Hay 10-12 segmentos (dependiendo del género de la
familia de los reovirus al que pertenezca el virión) (figura 22).
Hay diferencias significativas en el ciclo de vida de los miembros de las
familias reovirus y rotavirus. Dada su importancia clínica en humanos, nos
enfocaremos en los rotavirus.
|
 Figura 23 Rotavirus (Una partícula de cápside doble (izq.), y una cápside
interna única (der.))
Derecho reservado
Dra. Linda Stannard,
Universidad de Cape Town, Sudáfrica
Figura 23 Rotavirus (Una partícula de cápside doble (izq.), y una cápside
interna única (der.))
Derecho reservado
Dra. Linda Stannard,
Universidad de Cape Town, Sudáfrica |
ROTAVIRUS
(rota
= rueda (por la apariencia del virión bajo el microscopio de electrones)) (figura
23)
FIJACIÓN,
PENETRACIÓN Y PÉRDIDAD DE LAS ENVOLTURAS
Aún no está claro que es lo que ocurre exactamente
in vivo. Parece haber
una necesidad de que una proteasa remueva algo de la capa externa de la cápside
y genere una “partícula sub-viral intermedia” (PSVI) antes de que el virus pueda
entrar al citoplasma. In vivo, las PSVI son probablemente generadas por
digestión vía una en el tracto gastrointestinal. Una proteína de fijación viral
se expone en la PSVI, probablemente cerca de los vértices, y se une a receptores
de la célula huésped. La PSVI activada entra al citoplasma directamente o vía
endocitosis. En el citoplasma, el ARN del virión es copiado por la ARN
polimerasa viral mientras aún está en la nucleocápside que tienen menos
proteínas asociadas que una PSVI o que un virión.
TRANSCRIPCIÓN Y TRADUCCIÓN
Un ARN de cadena doble no funciona como un ARNm y por tanto el paso inicial es
la síntesis de ARNm (transcripción).
Los ARNm son sintetizados por ARN polimerasas codificadas por el virus mismo y
que vienen empacadas en el virión. El ARN es chapado y metilado por las enzimas
de empacamiento del virión. Luego es expulsado desde los vértices de la cápside.
|
 Figura 24
Figura 24 |
Los ARN,
son traducidos y las proteínas virales resultantes se ensamblan para formar
una cápside inmadura. Los ARNm son empacados en la cápside inmadura y luego
son copiados dentro de la cápside en ARN de cadena doble. (Se desconoce cómo
el virus se asegura de que cada partícula adquiera una copia de los 11 ARN
diferentes) (Figura 24). Ahora más ARNm es hecho por las recién formadas
cápsides inmaduras.
ENSAMBLAJE
Más
proteínas son sintetizadas y eventualmente las cápsides inmaduras yeman
hacia el lumen del retículo endoplásmico. Al hacer esto, adquieren una
envoltura transitoria que se pierde a medida que maduran. Esta es una
característica peculiar de los rotavirus.
LIBERACIÓN
Probablemente ocurre por lisis celular.
NOTA: EL CICLO REPLICATIVO COMPLETO OCURRE EN EL CITOPLASMA
|
|
|
 Regreso a la sección de
Virologia
Microbiología e Inmunología on line
Regreso a la sección de
Virologia
Microbiología e Inmunología on line
 Regreso a Microbiología e Inmunología On line
Regreso a Microbiología e Inmunología On line
Esta página se modificó
recientemente en
Saturday, February 07, 2015
Mantenimiento de la pagina por
Richard Hunt
|

 Figura
1 Polio virus
© 1999
Dr. J-Y Sgro,
Centro de Biotecnología de la Universidad de Wisconsin, Fuentes de Gráficas
Moleculares. Usado con autorización.
Figura
1 Polio virus
© 1999
Dr. J-Y Sgro,
Centro de Biotecnología de la Universidad de Wisconsin, Fuentes de Gráficas
Moleculares. Usado con autorización.
 Figura 4 Adaptada de Schaechter et al., Mechanisms of Microbial Disease, 2a
Ed.
Figura 4 Adaptada de Schaechter et al., Mechanisms of Microbial Disease, 2a
Ed. Figura 6 Rabdovirus en una célula epitelial de un pez
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
Figura 6 Rabdovirus en una célula epitelial de un pez
©
Dennis Kunkel
Microscopy, Inc.
Usado con autorización
 Figura
7 Estructura de un rabdovirus típico
Figura
7 Estructura de un rabdovirus típico
 Figura 8
Figura 8 Figura 9
Figura 9
 Figura 10
Figura 10
 Figura 11 Paramixovirus ©
Dr. Linda Stannard,
Universidad de Cape Town, Sudáfrica (usado con autorización)
Figura 11 Paramixovirus ©
Dr. Linda Stannard,
Universidad de Cape Town, Sudáfrica (usado con autorización)
 Figura
12 Estructura de un paramixovirus típico
Figura
12 Estructura de un paramixovirus típico Figura 13
Figura 13 Figura 14
Figura 14 Figura 15
Figura 15
 Figura 19 Estructura de un ortomixovirus típico
Figura 19 Estructura de un ortomixovirus típico Figura 20
Figura 20 Figura 21 Virión de un reovírus de mamífero
Deerechos reservados 1999
Dr. J-Y Sgro
– Centro de Biotecnología de la Universidad de Wisconsin, Reserva de
Gráficas Moleculares
Figura 21 Virión de un reovírus de mamífero
Deerechos reservados 1999
Dr. J-Y Sgro
– Centro de Biotecnología de la Universidad de Wisconsin, Reserva de
Gráficas Moleculares Figura 22 Estructura de un reovirus típico
Adaptado de Joklik et al. Zinsser Microbiology 2Oava. Ed.
Figura 22 Estructura de un reovirus típico
Adaptado de Joklik et al. Zinsser Microbiology 2Oava. Ed. Figura 23 Rotavirus (Una partícula de cápside doble (izq.), y una cápside
interna única (der.))
Derecho reservado
Dra. Linda Stannard,
Universidad de Cape Town, Sudáfrica
Figura 23 Rotavirus (Una partícula de cápside doble (izq.), y una cápside
interna única (der.))
Derecho reservado
Dra. Linda Stannard,
Universidad de Cape Town, Sudáfrica Figura 24
Figura 24





