|
x |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGY |
IMMUNOLOGY |
MYCOLOGY |
PARASITOLOGY |
VIROLOGY |
|
VIDEO LECTURE |
IMMUNOLOGIE -
CHAPITRE SIX
GENETIQUE DES
IMMUNOGLOBULINES
Gene Mayer,
Ph.D.
Emertius Professor of Pathology, Microbiology and Immunology
University of South Carolina
Denis
Hudrisier, Ph.D.
Centre national de la recherche scientifique (CNRS) · Institute of
Pharmacology and Structural Biology
Université de Toulouse
|

 |
|
EN ANGLAIS |
|
EM PORTUGUES - PORTUGUESE |
|
SHQIP - ALBANIAN |
Let us know what you think
FEEDBACK |
|
SEARCH |
|
|

 |
|
Logo image
© Jeffrey Nelson, Rush University, Chicago, Illinois and
The MicrobeLibrary |
|
|
LECTURE
Male et al. Immunology
7ème édition pp 80-85
Murray, et al. Medical Microbiology
5ème édition, pp 113-114
|
|
OBJECTIFS DU COURS
Décrire l’organisation et l’expression des familles de gènes des
immunoglobulines.
Expliquer les origines de la diversité des anticorps.
|
HISTORIQUE
Les données de séquençage des
acides aminés ont révélé qu’une région C donnée pouvait être retrouvée
associée à différentes régions V possibles. En outre, il a été montré qu'un
idiotype unique pouvait être retrouvé associé à différentes régions C
possibles (par exemple régions C d’IgM ou d’IgG). Pour expliquer ces données,
il a été proposé que les deux régions de la molécule d'immunoglobuline
pourraient avoir été codées par des gènes distincts et que les gènes codant
pour les régions V et C auraient été, en quelque sorte, joints avant qu’une
molécule d'immunoglobuline ne soit produite (soit deux gènes codant pour un
seul polypeptide). Ce concept alors révolutionnaire s’est finalement avéré
exact notamment grâce à l'avènement des technologies de recombinaison de
l'ADN. Les chaînes lourdes et légères d'immunoglobuline sont en effet codées
par trois familles de gènes présentes chacune sur un chromosome séparé : une
famille de gène code pour la chaîne lourde et une pour chacun des types de
chaînes légères. Chacune de ces familles de gènes possède plusieurs gènes
codant pour la région V et un ou plusieurs gènes codant pour la région C.
Les gènes codant pour la région V et la région C ne sont cependant pas
immédiatement adjacents les uns aux autres.
|
MOTS-CLÉS
Gène V
Gène C
Région J
Région D
Séquence Leader
Enhancer
Promoteur
Diversité des anticorps
Théorie germinale
Théorie des mutations somatiques
Insertions des nucléotides N
Diversité jonctionnelle
Diversité combinatoire
Multispécificité
Sélection clonale
|
FAMILLE DES GÈNES DE LA
CHAINE LEGERE
Organisation germinale
L’organisation germinale des gènes codant pour les chaînes légères kappa et
lambda dans des cellules indifférenciées est représentée dans la Figure 1.
-
Chaînes légères Lambda
La famille des gènes codant pour la chaîne légère lambda est composée de
4 gènes codant pour la région C, chacun codant pour un sous-type de
chaîne lambda, ainsi que d’environ 30 gènes codant pour la région V.
Chaque gène codant pour la région V est composé de deux exons, un (noté
L) code pour la séquence « Leader » et l’autre (V) qui code pour la plus
grande partie de la région variable. En amont de chacun des gènes C se
trouve un exon supplémentaire appelé J (pour jonction). Les exons L, V,
J et C sont séparés par des introns (séquences non-codantes).
-
Chaînes légères Kappa
La famille des gènes codant pour la chaîne légère kappa est composée
d’un seul gène codant pour la région C puisqu’il n’y a qu’un seul type
de chaîne kappa. Il y a un grand nombre de gènes de région V (environ
250 gènes), chaque gène codant pour la région V possédant un exon L et
un exon V. Dans la famille des gènes κ, il y a plusieurs exons J situés
entre les gènes V et C. Tous ces exons sont séparés par des introns.
|
 Figure 1
Figure 1
Organisation germinale des gènes codant pour les chaînes légères kappa
et lambda
|
Réarrangement des gènes et
expression
Au fur et à mesure qu’une cellule se différencie en lymphocyte B
mature et qu’une chaîne légère doit être assemblée, il se produit un
réarrangement de différents gènes (exons) et le gène commence à être
exprimé comme indiqué dans la Figure 2.
|
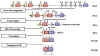 Figure
2 Figure
2
Lors de la différenciation d’une cellule en cellule B mature qui va
exprimer une chaîne légère, il y réaarrangement de différents gènes (exons).
|
Au fur et à mesure qu’une cellule
s'engage à devenir une cellule B assemblant une chaîne légère, il se produit
un réarrangement des gènes au niveau de l'ADN de telle sorte que l'un des
gènes V est positionné en regard de l'un des gènes J. Cela se produit par un
événement de recombinaison qui supprime les séquences présentes entre les
gènes V et J choisis. La sélection du gène V utilisé parmi les différentes
gènes V possible n'est pas totalement aléatoire : il y a une certaine
préférence pour l'utilisation de gènes V les plus proches de la région J.
Cependant, globalement, l'ensemble des gènes V peut être utilisé de telle
sorte que toutes les combinaisons de gènes V et J possibles peuvent être
générées.
Une conséquence de ce
réarrangement de l'ADN est que cela active la transcription du gène
réarrangé car un promoteur (P), qui est associé au gène V choisi, est
amené à proximité d'un amplificateur (E), qui est situé dans l'intron
entre les régions J et C. Suite à l’amorçage de la transcription à
partir du promoteur, un pré-ARNm est synthétisé à partir des séquences
contenant les régions L, C et VJ ainsi que des séquences d'introns entre
L et V et entre J et C (figure 2). Ce pré-ARNm est alors maturé (épissé)
dans le noyau et les introns restants sont éliminés. L'ARNm résultant
possède les exons L, VJ et C contigus.
L'ARNm est traduit dans le cytoplasme et la séquence L est éliminée lors
du transport de la protéine en cours d’élongation dans la lumière du
réticulum endoplasmique. La chaîne légère est assemblée avec une chaîne
lourde dans le réticulum endoplasmique et l'immunoglobuline est alors
sécrétée par la voie normale de sécrétion de protéines. Au final, la
région V de la chaîne légère mature est codée par la séquence des gènes
de la région V et J choisis et la région C, par la séquence du gène C.
|
 Figure 3
Figure 3
En plus des multiples exons J, les gènes de chaîne lourde contiennent
également plusieurs exons additionnels appelés exons D (diversité). Tous
les exons sont séparés par des introns.
|
FAMILLE DES GENES DES
CHAINES LOURDES
Organisation
germinale des gènes
L’organisation germinale des gènes codant pour les chaînes lourdes est
représentée dans la Figure 3.
Dans la famille des gènes
des chaînes lourdes, il existe de nombreux gènes C, un pour chaque
classe et sous-classe d’immunoglobuline. Chaque gène C est en fait
constitué de plusieurs exons codant pour les différents domaines CH
et pour la région charnière. Dans la famille des gènes codant pour
les chaînes lourdes, il y a également de nombreux gènes codant pour
les régions V, chacun étant composé d’un exon L et d’un exon V. En
plus, on retrouve plusieurs exons J et, également, spécifiquement
dans la famille de gènes codant pour les chaînes lourdes, on
retrouve plusieurs exons codant pour des régions D (Diversité). Tous
ces exons sont séparés par des introns comme cela est représenté
dans la Figure 3.
|
| |
Réarrangement des gènes et
expression
Au fur et à mesure qu’une cellule se différencie en lymphocyte B
mature et qu’une chaîne lourde doit être assemblée, il se produit un
réarrangement de différents gènes (exons) et le gène commence à être
exprimé comme indiqué dans les Figures 4 and 5.
|
 Figure 4
Figure 4
Au cours de l’initiation de la transcription à partir du promoteur, un
pré-ARNm est formé contenant les séquences des régions L, V, D, J Cμ et
Cδ ainsi que les séquences codant pour les introns placés entre L et V,
entre J et Cμ, et entre Cμ et Cδ
 Figure 5
Figure 5
Le pré-ARNm est maturé (épissé) dans le noyau et les introns restants,
incluant ceux présents entre les exons des gènes C sont éliminés.
|
Au fur et à mesure qu’une cellule
s'engage à devenir une cellule B assemblant une chaîne lourde, il se produit
deux réarrangements successifs au niveau de l'ADN. En premier lieu, l'une
des régions D est positionnée en regard de l'une des régions J puis l'un des
gènes V est amené en regard de la région DJ réarrangée. Cela se produit par
deux événements de recombinaison qui éliminent les séquences présentes entre
les régions V, D et J. Comme pour les chaînes légères, la sélection du gène
de la chaîne lourde V n'est pas totalement aléatoire mais, finalement, tous
les gènes V peuvent être utilisés.
Une conséquence de ces
réarrangements de l'ADN est l’activation de la transcription du gène car un
promoteur (P), qui est associée au gène V, est amené à proximité d'un
amplificateur (E), qui est situé dans l'intron entre les régions J et Cmu .
L’amorçage de la transcription à partir du promoteur conduit à la formation
d'un pré-ARNm à partir des séquences contenant les régions L, V, D, J Cmu et
Cdelta ainsi que les séquences introniques placées entre L et V, entre J et
Cmu, et entre Cmu et Cdelta (Figure 4).
L'ARNm est maturé (épissé) dans le
noyau et les introns restants, y compris ceux placés entre les exons des
gènes C, sont supprimés (voir figure 5). Le pré-ARNm peut alors être traduit
de deux manières : dans l'une, le bloc VDJ est placé à côté du gène Cmu. Les
ARNm résultants auront leurs exons L, V, D, J et CMU contigus permettant la
production d’une chaîne lourde de type mu. Dans le deuxième, le bloc VDJ est
placé à côté du gène Cdelta. Les ARNm résultants auront alors leurs exons L,
V, D, J et Cdelta contigus permettant la production d’une chaîne delta.
Les ARNm sont ensuite traduits
dans le cytoplasme et la séquence Leader est enlevée lorsque la protéine en
cours d’élongation est transportée dans la lumière du réticulum
endoplasmique. La chaîne lourde est assemblée avec une chaîne légère dans le
réticulum endoplasmique et l'immunoglobuline est sécrétée par la voie
normale de sécrétion de protéines. La région V de la chaîne lourde mature
est donc codée par des séquences du gène V, des régions D et J et la région
C par des séquences du gène C.
|
 Figure 6
Figure 6
De part et d’autre des exons V, J et D exons, on retrouve des séquences
particulières appelées « séquences signal de recombinaison » (RSS) , qui
fonctionnent par recombinaison. Chaque RSS consiste en un nonamère
conservé et un heptamère conservé séparés par 12 ou 23 paires de bases.
|
MECANISME DES
REARRANGEMENTS DE L’ADN
De part et d’autre des exons
V, J et D, il existe des séquences uniques appelées séquences signal de
recombinaison (RSS), qui fonctionnent en se recombinant. Chaque RSS est
constituée d'un nonamère conservé et un heptamère conservé qui sont
séparés par 12 ou 23 paires de bases (pb), comme illustré dans la figure
6. Les espaceurs de 12 pb et de 23 pb correspondent à une ou deux tours
de l'hélice d'ADN respectivement. La recombinaison se produit uniquement
entre une séquence signal présente sur 1 tour d’hélice avec une séquence
signal portée par 2 tours d’hélice. Dans le cas des chaînes légères λ on
trouve une séquence signal portée par 1 tour d’hélice en amont de l'exon
J et un signal porté par 2 tours d’hélice en aval de Vlambda. Dans le
cas des chaînes légères κ, il y a un signal porté par 1 tour d’hélice en
aval du gène Vkappa et un signal porté par 2 tours d’hélice en amont de
l'exon J. Dans le cas des chaînes lourdes, il y a des séquences signal
portées par 1 tour d’hélice de chaque côté de l'exon D et des séquences
signal portées par 2 tours d’hélice en aval du gène V et en amont de
l'exon J. Cette organisation garantit que des événements de
recombinaison corrects vont se produire entre les bons exons.
Les événements de
recombinaison conduisent à la suppression des introns placés entre les
exons V et J recombinés dans le cas des chaînes légères ou entre les
exons V, D et J recombinés dans le cas des chaînes lourdes. L'événement
de recombinaison est catalysée par deux protéines, Rag-1 et Rag-2. Des
mutations dans les gènes codant pour ces protéines entraîne une
immunodéficience grave appelée severe combined immunodeficiency disease
(dans laquelle à la fois les cellules T et B sont absentes), puisque ces
protéines et les RSS sont impliquées dans la génération des récepteurs à
l’antigène non seulement des cellules B mais aussi des cellules T.
|
 Figure 7 Ordre dans l’expression des gènes d’immunoglobulines: cas de la
chaîne lourde
Figure 7 Ordre dans l’expression des gènes d’immunoglobulines: cas de la
chaîne lourde

Figure 8
Ordre dans l’expression des gènes d’immunoglobulines: cas de la chaîne
légère
|
ORDRE DE L’EXPRESSION DES GENES DANS LA
FAMILLE DES GENES D’IMMUNOGLOBULINES
Chaque cellule B individuelle produit un seul
type de chaîne légère et une seule classe de chaîne lourde (à l’exception de
la cellule B mature qui exprime à la fois une chaîne lourde μ et une chaîne
lourde δ mais la spécificité antigénique est la même car la même séquence
VDJ est associéesaux chaînes μ et δ). Dans la mesure où chaque cellule B
possède les chromosomes maternels et paternels pouvant chacun coder pour des
immunoglobulines, il doit y avoir une certaine organisation dans
l’expression des gènes permettant qu’un seul type de chaîne légère et une
seule classe de chaîne lourde soient produites.
L’ordre dans lequel les gènes
d’immunoglobulines sont exprimés dans une cellule B est indiqué dans les
Figures 7 et 8.
Chaîne lourde
(Figure 7)
La cellule tente d’abord de réarranger l’un de ses gènes de chaîne lourde :
dans certaines cellules, c’est le chromosome maternel qui est réarrangé
alors que, dans d’autres, c’est le chromosome paternel. Si le réarrangement
est productif, de telle sorte qu’une chaîne lourde est produite, alors les
autres réarrangements sont bloqués. A l’inverse, si la première tentative de
réarrangement est improductive (pas de chaîne lourde produite), la cellule
va alors tenter de réarranger les gènes de chaîne lourde sur l’autre
chromosome. Si la cellule ne parvient pas à réarranger ses gènes de chaîne
lourde, alors cette cellule est destinée à être éliminée.
|
| |
Chaine légère Kappa
(Figure 8)
Lorsque la cellule parvient à réarranger un gène de chaîne lourde,
elle tente alors de réarranger ses gènes codant pour une chaîne
légère kappa. De nouveau, au hasard, la cellule va tenter de
réarranger ses gènes de chaîne légère d’origine maternelle ou
paternelle. Si le réarrangement est improductif (c’est à dire que la
cellule ne produit pas de chaîne légère), alors la cellule tente de
réarranger ses gènes kappa sur l’autre chromosome. Si la cellule
réussit à réarranger ses gène de chaîne légère kappa, elle deviendra
une cellule B capable de produire une immunoglobuline possédant une
chaîne légère kappa.
|
| |
Lambda light chain
(Figure 8)
Si une cellule ne parvient pas à réarranger ses gènes de chaîne
légère kappa, elle essayera alors de produire une chaîne légère
lambda. De nouveau, au hasard, la cellule va tenter de réarranger
ses gènes de chaîne légère d’origine maternelle ou paternelle. Si le
réarrangement est improductif (c’est à dire que la cellule ne
produit pas de chaîne légère), alors la cellule tente de réarranger
ses gènes lambda sur l’autre chromosome. Si la cellule réussit à
réarranger ses gène de chaîne légère lambda, elle deviendra une
cellule B capable de produire une immunoglobuline possédant une
chaîne légère lambda.
La séquence organisée des réarrangements des gènes d’immunoglobuline
explique:
- Pourquoi une cellule B donnée ne
peut produire qu’un seul type d’immunoglobuline avec un seul
type de chaîne lourde et un seul type de chaîne légère.
- Pourquoi une cellule B donnée peut
seulement produire des anticorps possédant une seule spécificité.
- Pourquoi il y a exclusion
allélique des allotypes d’immunoglobuline au niveau d’une
molécule individuelle d’immunoglobuline mais expression co-dominante
des allotypes dans un organisme donné.
|
 Figure 9
Figure 9
Concepts actuels sur l’origine de la diversité des anticorps.
|
ORIGINE DE LA
DIVERSITÉ DES ANTICORPS
Bases de la
problématique
La diversité des anticorps fait référence à la somme de toutes les
spécificités d’anticorps possibles que peut produire un organisme.
Il est estimé que l’on peut produite 107 - 108 molécules d’anticorps
différentes par leur spécificité. Une des questions majeure de
l’immunologie est de savoir comment il est possible de produire
autant de molécules différentes. Les théories permettant d’expliquer
l’origine de la diversité des anticorps sont de deux ordres :
Théorie germinale
Cette théorie postule que nous disposons de gènes V différents
pour chaque anticorps possible fabriqué.
Théorie des mutations somatiques
Cette théorie postule que nous disposons seulement de quelques
gènes codant pour les régions V et que la diversité est générée
par des mutations somatiques qui se produisent au niveau des ces
gènes.
Concepts
actuels
Les concepts actuels donnent du crédit aux deux théories proposées.
On pense que la diversité des anticorps est générée par les
mécanismes suivants :
1. Un grand nombre de
gènes V
Il y a:
a) 30 gènes V
lambda
b) 300 gènes V kappa
c) 1000 gènes V de chaînes lourdes
2. Jonctions V-J et
V-D-J
La région codée par les gènes V et J de la chaîne légère et les
gènes V, D et J de la chaîne lourde est la troisième boucle
hypervariable (CDR3) de la région V de chacune de ces chaînes.
Comme les réarrangements V-J d’une part et V-D-J d’autre part se
font au hasard, il en résulte une grande diversité à la jonction
V-J et V-D-J.
3. Diversité
jonctionnelle (imprécisions aux sites de recombinaison V-J, V-D
et D-J) - (Figure 9)
|
| |
La recombinaison V-J et V-D-J
n’est pas toujours parfaite et une diversité additionnelle résulte
d’erreurs commises lors de la recombinaison qui rapproche les gènes V et
J ou encore D et J. Ces imprécisions triplent au moins le degré de
diversité obtenu par la simple diversité combinatoire V-J et V-D-J. La
diversité due à ce mécanisme se produit pour la région qui code pour la
troisième boucle hypervariable et affecte donc directement le site de
liaison de l’anticorps.
4. Région d’insertion de
nucléotides N
A la jonction entre les segments D et J on retrouve souvent une
insertion de quelques nucléotides, catalysée par une enzyme appelée «
terminal transférase ». La terminal transférase catalyse la
polymérisation au hasard de nucléotides dans l’ADN sans nécessité de
matrice. Cela conduit à davantage encore de diversité sur la troisième
région hypervariable.
5. Mutation somatique
Il a été démontré qu’il se produisait des mutations somatiques au niveau
des gènes V, notamment sur les séquences codant pour la seconde région
hypervariable (CDR2). Ces mutations contribuent dans une certaine mesure
à la diversité des anticorps.
6. Combinatoire d’association
Chaque cellule B individuelle a le potentiel pour produire n’importe
laquelle des chaînes lourdes et n’importe laquelle des chaînes légères.
Chaque chaîne légère peut également s’associer à chaque chaîne lourde ce
qui contribue à davantage de diversité.
7. Multispécificité
En raison des réactions croisées entre déterminants antigéniques de
structure similaire, un anticorps peut souvent réagir avec plus qu’un
seul antigène. C’est la multispécificité. La multispécificité contribue
à la diversité des anticorps.
Un exemple de comment ces
différents mécanismes peuvent générer un haut niveau de diversité
est illustré ci-dessous :
|
| |
|
|
Récepteur
à l’antigène des lymphocytes B (Immunoglobulines) |
|
Chaîne lourde |
Chaîne
légère Kappa |
|
segments de
gènes V |
1000 |
300 |
|
segments de
gènes D |
15 |
- |
|
segments de
gènes J |
4 |
4 |
|
Insertion de
nucléotides N |
++ |
- |
|
Diversité
jonctionnelle |
+++ |
+ |
|
Mutation
somatique |
+ |
+ |
|
Diversité
combinatoire |
V x D x J
1000 X 15 X 4 |
V x J
300 x 4 |
|
Total |
6 x 104 |
1.2 x 103 |
|
|
|
|
Combinatoire
des appariements chaîne légère/chaîne lourde |
7.2 x 107 |
|
| |
Ces calculs ne prennent
pas en considération les contributions des chaînes légères lambda,
la diversité jonctionnelle, les mutations somatiques, les insertions
de nucléotides N ou la multispécificité.
Le processus de réarrangement des gènes des chaînes lourdes et
légères et l'association combinatoire de ces chaînes se produisent
au cours du développement des cellules B : ces étapes sont donc
indépendantes de l'antigène. Des clones de cellules B exprimant
toutes les spécificités d'anticorps possibles sont produits au cours
du développement et, après son introduction, l'antigène sélectionne
simplement les clones qui ont le récepteur approprié. Les clones
sélectionnés sont alors activés, prolifèrent et se différencient en
cellules sécrétant des anticorps plasmatiques.
LE RECEPTEUR A L’ANTIGENE
DES CELLULES T
Les cellules T expriment
également un récepteur pour l'antigène à leur surface. Ce récepteur
n'est pas une molécule d'immunoglobuline, mais il est composé de
deux chaînes polypeptidiques différentes qui ont des régions
constantes et variables analogues aux immunoglobulines. La diversité
des récepteurs à l’antigène des cellules T est également générée de
la même manière que celle décrite pour la diversité des anticorps
(par exemple par assemblage de segments de gènes VJ et VDJ et leur
combinatoire d'association). Cependant, aucune mutation somatique
n’a été observée dans le cas des cellules T.
|
|


|
 Retourner à la section d'immunologie de Microbiologie et Immunologie On-line
Retourner à la section d'immunologie de Microbiologie et Immunologie On-line
This page last changed on
Tuesday, November 27, 2012
Page maintained by
Richard Hunt
Please report any problems to
richard.hunt@uscmed.sc.edu
|

 Figure
2
Figure
2 Figure 3
Figure 3 Figure 4
Figure 4 Figure 6
Figure 6 Figure 7 Ordre dans l’expression des gènes d’immunoglobulines: cas de la
chaîne lourde
Figure 7 Ordre dans l’expression des gènes d’immunoglobulines: cas de la
chaîne lourde
 Figure 9
Figure 9

