|
xx |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGIE |
IMMUNOLOGIE |
MYCOLOGY |
PARASITOLOGY |
VIROLOGIE |
|
VIDEO
LECTURES |
Virologie Chapitre 5
La génétique des virus
Dr Margaret Hunt
University of South Carolina School of Medicine
Columbia SC
USA
Dr Dorian McIlroy
Université de Nantes
France
|
|
En
Español |
|
EN ANGLAIS |
|
NË SHQIPTARE |
Let us know what you think
FEEDBACK |
|
SEARCH |
|
|

 |
|
|
|
|
|
|
Généralités
Les virus se répliquent rapidement, et normalement il y a un
grand nombre de particules virales produites à partir d’une cellule infectée.
Les virus ont donc un plus grand potentiel que les organismes cellulaires à
générer des mutations dans un laps de temps court .
Les génomes viraux peuvent être modifiés par la mutation ou par
la recombinaison.
La nature du matériel génétique (ARN ou ADN, segmenté ou monocaténaire) joue un
rôle important dans la génétique des virus, et la fréquence relative de la
mutation et de la recombinaison.
Virus Mutants
Origine des Mutations
Mutations spontanées
Ce type de mutation se produit lors de la réplication virale, par exemple à
cause des erreurs de copies introduites par la polymérase responsable de la
réplication du génome, ou par l’incorporation de tautomères des bases
nucléotidiques.
Les virus à ADN ont tendance à plus de stabilité génétique
que les virus à ARN ou des rétrovirus. Il existe des mécanismes de
correction d’erreurs pour la réparation de l’ADN chez la cellule hôte, mais
pas de système comparable pour l’ARN.
Certains virus à ARN ont des génomes remarquablement
stables. Il est probable que ces virus subissent le même taux de mutation
que d’autres virus à ARN, mais qu’ils sont tellement bien adaptés pour
maximiser leur transmission et réplication, que même l’incorporation d’un
faible nombre de mutations rendrait les virus mutants moins efficace que le
virus sauvage (wild-type, ou WT). Dans ce cas, on peut considérer que la
stabilité génétique d’un virus à ARN est un résultat de son adaptation à un
hôte possédant un grand génome en ADN, qui reste constante à l’échelle de
temps de la réplication virale.
Mutations Induites
Des traitements chimiques et physiques peuvent induire des
mutations dans des acides nucléiques
Mutagènes chimiques
Composés agissant directement sur les bases
nucléotidiques - par exemple, l’acide nitreux, ou les espèces actives
dérivées d’oxygène (EADO).
Composés avec une action indirecte – par exemple, les
analogues des bases nucléotidiques, comme le 5-bromouracile, qui peuvent
s’insérer dans l’ADN ou l’ARN et qui s’apparient plus facilement avec
une base incorrecte.
Mutagènes physiques
Les rayonnements UV ou les rayons-X. Il est important de
noter, par contre, que la plupart des effets mutagènes des rayons-X est
une conséquence de la génération des EADO à proximité de l’ADN.
Types de Mutation
Les mutations peuvent être ponctuelles (une base remplacée par
une autre) ou des insertions/délétions de un ou de plusieurs nucléotides.
Exemples de phénotypes
"mutants" chez les virus
Phénotype = propriétés observables d’un organisme
Mutants "Conditional Lethal"
Ces mutants se répliquent sous certaines conditions, mais
pas sous d’autres. En contraste, la virus sauvage se réplique sous les deux
conditions.
1) Mutants thermosensibles (ts) – le virus mutant se
réplique chez des cellules cultivées à basse température (exemple 31°C)
mais pas chez des cellules cultivées à haute température (exemple 39°C).
La souche sauvage se réplique à 39°C et 31°C. Dans ce cas, il semble que
la protéine virale touchée par la mutation est incapable de maintenir
une conformation fonctionnelle à la température élevée.
2) Spectre d’hôte – le virus mutant est capable de se
répliquer uniquement chez une partie des types cellulaires qui
supportent la réplication du virus sauvage. Ce type de mutant est utile
dans la recherche sur le rôle des facteurs de l’hôte dans la réplication
virale.
Mutants de taille de plage de lyse
Les plages de lyse formées par le virus mutant ont une
taille anormale – elles sont soit plus grandes, soit plus petites que
les plages de lyses formées par le virus sauvage. La pathogénicité de ce
type de mutant est parfois modifiée – un virus mutant qui se réplique
moins bien produira des petites plages de lyse, et peut s’avérer moins
virulent.
Résistance aux antiviraux
La réplication du virus mutant n’est plus inhibée par
des médicaments antiviraux. La sélection de ce type de mutant au cours
de la thérapie antivirale est une possibilité qui doit être prise en
compte en clinique.
Mutants déficients en une enzyme
virale
Parmi les différentes enzymes virales, certaines ne sont
pas toujours essentielles à la réplication virale. Par exemple, chez le
virus Herpes simplex 1, la thymidine kinase n’est pas nécessaire à la
réplication du virus en culture cellulaire, mais elle est importante
pour l’infection des neurones in vivo. Il est donc possible d’isoler des
virus déficients en thymidine kinase en culture cellulaire.
Mutants "chauds"
Ces mutants se répliquent mieux que le virus sauvage à
température élevée. Les mutants "chauds" peuvent s’avérer plus
pathogènes que le virus sauvage, car la réplication d’un mutant "chaud"
ne sera pas inhibée par la fièvre chez l’hôte.
Mutants atténués
Tous les mutants qui provoquent des symptômes moins
sévères que le virus sauvage, ou qui résultent en une infection
asymptomatique sont des mutants atténués. Plusieurs vaccins sont des
virus atténués, et l’étude des mutations responsables de l’atténuation
des symptômes aide à comprendre les mécanismes de pathogénèse chez le
virus sauvage.
|
 Fig. 1. Recombinaison par "copy choice"
Fig. 1. Recombinaison par "copy choice" |
Echange du matériel génétique Les virus ne se reproduisent
pas par voie sexuée, et les acides nucléiques de deux particules virales ne
peuvent pas être échangés directement (à la manière de la conjugaison
bactérienne). Néanmoins, les échanges génétiques entre virus peuvent avoir lieu,
dans le cas où plusieurs particules virales infectent la même cellule. Au sein
de cette cellule infectée, les génomes des différents virus peuvent se
rencontrer, et éventuellement se mélanger, en employant deux mécanismes
distincts :
La recombinaison classique
La recombinaison implique la cassure d’un brin d’acide nucléique,
l’échange d’acide nucléique entre deux génomes viraux, et la religation de
la cassure du brin d’acide nucléique. Ce mécanisme "cassure-soudure" de
recombinaison est fréquent chez les virus à ADN et chez les rétrovirus. Il
est facilité par la présence d’enzymes de l’hôte capables d’effectuer la
recombinaison de l’ADN. Par contre, ce type de recombinaison est très rare
chez les virus à ARN, car il n’existe pas d’enzymes cytoplasmiques de l’hôte
capables de catalyser la recombinaison de l’ARN.
Chez les Picornaviridae, une forme de recombinaison à faible efficacité a
lieu. Le mécanisme semble consister en un changement de matrice en cours de
la transcription de l’ARN génomique par l’ARN polymérase virale (mécanisme
dit "copy choice", Figure 1.)
La recombinaison génétique est fréquente chez les Coronaviridae , qui
comme les Picornaviridae ont un génome en ARN brin (+). Encore une fois, le
mécanisme de recombinaison dépend de l’ARN polymérase virale, et n’est pas
semblable au mécanisme de recombinaison de l’ADN.
Jusqu’à présent il n’y a pas d’indication que la recombinaison génétique
se produit chez les virus à ARN brin (-). Chez ces virus, l’ARN génomique
reste associé à la nucléocapside pendant la réplication du génome. Il n’est
donc pas disponible pour l’appariement de bases, qui est nécessaire à la
recombinaison.
La recombinaison permet aux virus d’assimiler de l’information génétique
des virus du même genre ou famille, et parfois d’un virus totalement
différent. Il est également possible (notamment chez les Retroviridae) pour
un virus d’incorporer de l’ADN venant du génome de la cellule hôte.
|
 Fig.
2. "Marker Rescue", ou récupération de trait Fig.
2. "Marker Rescue", ou récupération de trait |
Exploitation technique de la recombinaison
chez les virus
a) Cartographie génétique – plus la distance entre deux gènes est grande,
plus la recombinaison sera fréquente entre eux.
b) Le génome d’un virus mutant peut être "réparé" par recombinaison
avec des fragments d’ADN provenant d’un virus sauvage. Ce processus
conduit à la transformation d’un virus avec un phénotype mutant en un
virus sauvage, et permet d’associer le phénotype du mutant avec une
région du génome viral. Cette technique s’appelle "marker rescue" (littéralement,
secours, ou récupération de trait) car en quelque sorte le virus mutant
est "secouru" par l’ADN du virus sauvage. La même technique perme
|
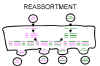 Fig. 3. Réassortiment des génes chez les virus à génome segmenté
Fig. 3. Réassortiment des génes chez les virus à génome segmenté |
Le réassortiment Si un
virus possède un génome segmenté, et si deux variantes de ce virus infectent
la même cellule , les particules virales libérées par la cellule infectée
peuvent comporter un génome mosaïque, certains segments venant de l'un des
virus parents, d’autres segments venant de l’autre virus.
Il s'agit d'un processus efficace - mais il est limitée à des virus dont le
génome est segmenté - jusqu'ici les seuls virus humains caractérisés avec
des génomes segmentés sont des virus à ARN, en particulier les
Orthomyxoviridae, les Reoviridae, les Arenaviridae, et les Bunyaviridae.
Le réassortiment joue un rôle important dans la nature dans l’émergnece de
nouveaux virus réassortants. Il est également utile dans des expériences au
laboratoire (figure 3), par exemple, dans l'attribution des fonctions aux
différents segments du génome. Par exemple, dans un virus réassortant, si un
segment provient de virus A et le reste de virus B, si le virus réassortant
possède le même sérotype que le virus A, on pourra conclure que le segment
provenant du virus A code pour une protéine immunogène à la surface du virus
– soit une glycoprotéine de surface, soit une protéine de capside, dans le
cas d’un virus nu.
Le réassortiment est un mécanisme non-classique de la recombinaison qui
n’impliquer pas de cassure de l’acide nucléique du génome viral.
|
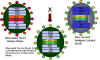 Fig. 4. Réassortiment des génes entre une souche atténuée du virus de la
grippe et une nouvelle souche virulente lors de l'élaboration d'un
vaccin atténué contre la grippe. Adapté à partir de Treanor JJ Infect.
Med. 15:714
Fig. 4. Réassortiment des génes entre une souche atténuée du virus de la
grippe et une nouvelle souche virulente lors de l'élaboration d'un
vaccin atténué contre la grippe. Adapté à partir de Treanor JJ Infect.
Med. 15:714 |
Applications de la génétique virale
L’un des vaccins contre la grippe, le FluMist (autorisé aux Etats-Unis
depuis 2003, depuis 2011 en Europe) a été élaboré en exploitant certains des
principes évoqués ci-dessus. Le vaccin est trivalent, c’est-à-dire qu’il
contient trois souches de virus de la grippe (deux souches grippe A, et une
souche de grippe B).
Chacun des trois souches qui composent le vaccin est un virus réassortant
entre un virus mutant adapté au froid, et un virus sauvage. Les segments
géniques codant pour les protéines de surface du virus de la grippe (HA -
hémagglutinine et NA - neuraminidase) proviennent du virus sauvage, et les
six autres segments génomiques proviennent du virus adapté au froid. Les
souches vaccinales possèdent donc les propriétés antigéniques des virus
sauvages, mais en sont beaucoup moins pathogènes, car leur réplication est
optimale à 25 °C, plutôt qu’à 37°C. Ainsi ils peuvent se développer dans les
voies respiratoires supérieures, où il fait plus frais, mais se répliquent
mal dans le tractus respiratoire inférieur, qui est plus chaud. L’adaptation
au froid est dû à de multiples modifications dans les différents segments du
génome (en dehors des segments codant pour la HA et la NA), et la réversion
vers un phénotype virulent est donc peu probable.
Le FluMist est un vaccin "vivant" attenué administré par voie intranasale
sous forme de pulvérisation qui est capable d’induire une immunité mucosale
et systémique.
Deux vaccins réassortant "vivant" atténués sont actuellement disponibles
pour les rotavirus (RotaTeq de Merck, et Rotarix de GSK).
|
| |
Complémentation La
complémentation d’un phénotype mutant indique une interaction au niveau
fonctionnel, et non pas une échange au niveau des acides nucléiques. Par
exemple, si nous prenons deux mutants avec une lésion ts (sensible à la
température) dans des gènes différents, aucun des deux ne pourra se
répliquer à une température non-permissive (par exemple 39°C). Si nous
infectons la même cellule avec les deux virus mutants, chaque mutant peut
assurer la fonction manquante de l'autre et le cycle de réplication virale
pourra s’accomplir. Néanmoins, en l’absence de recombinaison génétique, les
virions libérés par la cellule doublement infectée contiendront encore des
génomes mutants ts et seront encore sensible à la température.
Il est possible d'utiliser la complémentation afin de regrouper et
cartographier des mutants ts, car mutants deux mutants ts dans le même gène
ne seront généralement pas en mesure de se compléter mutuellement. C'est un
outil de base en génétique pour déterminer si les mutations se trouvent au
sein d'un même gène, ou chez des gènes différents, et de déterminer le
nombre minimum de gènes distinctes qui contribuent à une fonction précise.
|
| |
Réactivation à forte MOI
Si un virus à ADN double brin est inactivé par irradiation ultraviolette, le
virus peut se "réactiver" si l'on infecte des cellules avec le virus
inactivé à une très forte multiplicité d'infection (MOI). Dans ce cas, un
grand nombre de particules virales par cellule rentrent dans chaque cellule
infectée, ce qui permet aux différents génomes inactivés de coopérer d'une
certaine manière.
Dans un premier temps, il est probable que la complémentation permette au
virus de se développer, puisque les gènes inactivés dans une particule
virale peuvent être encore fonctionnels dans l'un des autres. La forte MOI
permet de rassembler à l'intérieur d'une même cellule, un génome complet à
partir de plusieurs génomes défectueux. Comme le nombre de copies des
génomes viraux augmente à cause de la réplication virale, la recombinaison
peut se produire, entraînant de nouvelles permutations des différents
génotypes mutés, et ce processus permet, parfois, de régénérer le virus
sauvage. Les Virus Défectueux
Les virus défectueux ne possèdent pas la totalité des gènes nécessaires à un
cycle infectieux complet (en effet, beaucoup de virus défectueux ont
incorporés des délétions dans leur génome) - et ils ont donc besoin d'un
deuxième virus pour assurer les fonctions manquantes. Ce second virus est
appelé un virus auxiliaire.
Le génome d'un virus défectueux doit comprendre au minimum 1) les séquences
qui permettent une polymérase (virale ou cellulaire) de répliquer le génome
défectueux, et 2) une séquence d’empaquetage qui permet l'incorporation du
génome à l'intérieur de la particule virale. Certains virus défectueux sont
capables d'assurer plus de fonctions de façon autonome. Quelques exemples
de virus défectueux:
Certains rétrovirus ont incorporé des séquences provenant de la cellule hôte
dans leur génome au dépens des gènes viraux nécessaires pour leur
réplication. Ces virus ont besoin d'un virus étroitement apparentés, qui
conserve ces fonctions, comme virus auxiliaire. Certains virus qui sont
incapables de se répliquer de façon autonome peuvent utiliser des virus non-apparentés
comme virus auxiliaires. Par exemple, le virus de l'hépatite delta (un virus
à ARN) ne code pas pour sa propre protéine d'enveloppe, mais utilise celle
du virus de l'hépatite B (un virus à ADN). Dans de tels cas, le virus "non-autonome"
est le plus souvent considéré comme un virus satellite, plutôt qu'un virus
défectueux.
|
| |
Particules "Défectueuse
à Interférence" (DI)
La réplication du virus auxiliaire
peut être moins efficace en présence du virus défectueux qu'en son absence.
En effet, à l'intérieur de la cellule infectée, le génome du virus
défectueux est en concurrence avec celui du virus auxiliaire. Ce phénomène
est appelé "interférence", et les particules défectueuses responsables de ce
phénomène sont connus comme des particules "défectueux" à interférence (DI).
Tous les virus défectueux ne provoquent pas de l'interférence interfèrent,
mais il s'agit néanmoins d'un phénomène fréquent chez les virus défectueux.
Notez qu'il est possible que les
particules DI puissent moduler la pathologie des infections naturelles. Par
exemple, chez les virus des plantes, la co-infection par un virus satellite
est le plus souvent associée avec phytopathologie atténuée, par rapport avec
une infection par le virus auxiliaire tout seul. En revanche, la
co-infection par le virus de l'hépatite delta à tendance à exacerber la
pathologie des infections par le virus de l'hépatite B.
|
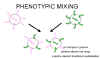 Fig. 5. Mélange phénotypique entre deux virus infectant la même cellule
Fig. 5. Mélange phénotypique entre deux virus infectant la même cellule
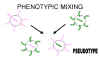 Fig. 6. Formation d'un virus pseudotypé par mélange phénotypique
Fig. 6. Formation d'un virus pseudotypé par mélange phénotypique |
Mélange phénotypique
Si deux virus différents infectent la même cellule, les
virus descendants peuvent contenir des composants d'enveloppe ou de capside
provenant de deux virus parents. C'est ce qu'on appelle le mélange
phénotypique (figure 5). Elle n'entraîne pas de modification dans le
matériel génétique, et la descendance de ces virions sera déterminée par le
génome à l'intérieure de la particule virale, et non par la nature de
l'enveloppe ou de la capside à l'extérieur de la particule.
Un mélange phénotypique peut se produire entre des virus apparentés, par
exemple deux membres différents de la famille des Picornaviridae, ou entre
des virus génétiquement distinctes, par exemple, entre un rhabdovirus et un
paramyxovirus. Dans ce dernier cas, les deux virus impliqués sont
généralement enveloppés, car il semble que les contraintes moléculaires
impliquées dans l'empaquetage de la nucléocapside d'un virus dans
l'enveloppe d'un autre soient moins stringentes que lorsque le génome d'un
virus est empaqueté dans la capside icosaédrique d'un autre virus.
Il est également possible de produire des particules chez lesquelles
l'enveloppe est entièrement dérivée d'un autre virus. Par exemple, la
nucléocapside d'un rétrovirus peut être associée avec l'enveloppe d'un
rhabdovirus. Ce genre de mélange phénotypique est souvent appelé un
pseudotype, et de telles particules sont désignées de pseudovirions (figure
6). Le pseudotype décrit ci-dessus possèdent les caractéristiques
d'antigénicité, d'adsorption et de pénétration du rhabdovirus, et ensuite,
lors des étapes intracellulaires du cycle de réplication, il se comportera
comme un rétrovirus. Les particules libérées à la fin du cycle de
réplication seront des rétrovirus "normaux", sans l'apport de l'enveloppe du
rhabdovirus. Il en résulte que les pseudotypes sont dotés d'un tropisme hôte
modifié de façon temporaire, qui dure uniquement pendant le premier cycle de
réplication. Si le génome rétroviral ne code pas pour une glycoprotéine
d'enveloppe, le virus pseudotypé ne pourra assurer qu'un seul cycle de
réplication.
|
|
|
 Retour à la section de virologie de Microbiologie et Immunologie On-line
Retour à la section de virologie de Microbiologie et Immunologie On-line
This page last changed on
Wednesday, February 19, 2014
Page maintained by
Richard Hunt
|

 Fig. 3. Réassortiment des génes chez les virus à génome segmenté
Fig. 3. Réassortiment des génes chez les virus à génome segmenté Fig. 4. Réassortiment des génes entre une souche atténuée du virus de la
grippe et une nouvelle souche virulente lors de l'élaboration d'un
vaccin atténué contre la grippe. Adapté à partir de Treanor JJ Infect.
Med. 15:714
Fig. 4. Réassortiment des génes entre une souche atténuée du virus de la
grippe et une nouvelle souche virulente lors de l'élaboration d'un
vaccin atténué contre la grippe. Adapté à partir de Treanor JJ Infect.
Med. 15:714 Fig. 5. Mélange phénotypique entre deux virus infectant la même cellule
Fig. 5. Mélange phénotypique entre deux virus infectant la même cellule
