|
x |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGIE |
IMMUNOLOGIE |
MYCOLOGY |
PARASITOLOGY |
VIROLOGIE |
|
VIDEO LECTURE |
VIROLOGIE - CHAPITRE TROIS
Stratégies de Réplication des virus à ADN
Dr Margaret Hunt
University of South Carolina School of Medicine
Columbia SC
USA
Dr Dorian McIlroy
Université de Nantes
France
|
|
EN ANGLAIS |
|
En
Español |
|
NË SHQIPTARE |
Let us know what you think
FEEDBACK |
|
SEARCH |
|
|
|
|

 |
|
Logo image © Jeffrey
Nelson, Rush University, Chicago, Illinois and
The MicrobeLibrary |
|
READING:
Murray et al., Microbiology
6th Ed |
|
|
Rappel - Généralités
Les génomes viraux contiennent de l’information qui:
-
assure la réplication du génome viral
-
assure l'empaquetage du génome viral dans des particules
composées de protéines virales
-
modifie la structure et / ou la fonction de la cellule hôte
afin de favoriser la réplication virale
Stratégies de réplication virale
Le terme "stratégie" de réplication virale fait référence à la manière dont
chaque virus effectue les opérations décrites ci-dessus. Puisqu’un virus est un
parasite intracellulaire, il doit opérer à l’intérieur des limites imposées par
la cellule hôte, ou réussir à contourner ces limitations.
|
| |
Stratégies de réplication
de virus à ADN
Généralités
-
Tout virus doit obligatoirement
synthétiser des ARN messagers (ARNm) qui peuvent être traduits en
protéine par les ribosomes de la cellule hôte.
-
Tout virus doit répliquer son génome.
-
Les enzymes de l'hôte impliquées dans
la synthèse de l'ARNm et dans la réplication de l'ADN sont nucléaires (à
l'exception de celles de la mitochondrie). Ainsi, si un virus "veut"
exploiter ces enzymes, il doit pénétrer dans le noyau.
|
| |
Famille Parvoviridae
Les Parvoviridae sont dotés d'une capside icosaédrique nue, de très petite
taille (diamètre de 18 à 25nm; parvum=petit), qui entoure un génome en ADN
simple brin. La réplication de l'ADN génomique a lieu dans le noyau.
Un grand nombre de Parvoviridae humains sont des virus satellites qui
sont dépendants d'une co-infection par un autre virus à ADN – soit un
Adenovirus, soit un Herpesvirus – pour compléter leur cycle de réplication.
Ces Dependovirus (aussi dénommés "Adeno-Associated viruses" ou AAV) ont été
développé en tant vecteurs de thérapie génique. En revanche, les bocavirus,
qui provoquent des infections respiratoires, et le parvovirus humain B-19
sont capables de se répliquer sans l'aide d'un virus auxiliaire.
Le parvovirus B-19 se réplique chez des cellules qui sont en train de se
diviser – principalement les cellules progénitrices des érythrocytes dans la
moelle osseuse – et provoque la "cinquième maladie", erythema infectiosum.
La pathologie de cette maladie est généralement bénigne, mais la
perturbation de l’érythropoïèse qui en résulte peut provoquer une anémie
aiguë chez des patients atteints de différents types d’anémie hémolytiques
sévères.
Attachement, entrée et décapsidation
Le parvovirus B-19 se fixe sur l'antigène P des érythrocytes, qui est
également présent à la surface des précurseurs des globules rouges. La
fixation du virion sur l'antigène P est suivi de son endocytose provoqué
par l'intégrine 51, qui est exprimée par les précurseur des
érythrocytes, mais pas par les hématies. L'utilisation de cet intégrine
comme co-récepteur pour l'entrée du virus garantit que le parvovirus
B-19 ne rentre pas dans les hématies, qui sont incapables d'assurer la
réplication du virus, vu qu'elles sont dépourvues de noyau.
Après l'entrée, l'endolysosome est perturbé par la protéine mineure
de capside, VP1, et la particule virale échappe dans le cytoplasme. Les
virions sont ensuite acheminés vers le noyau par le réseau de
microtubules. Ils sont suffisamment petits pour passer à travers les
pores nucléaires, et la décapsidation a lieu à l'intérieur du noyau.
Expression des gènes viraux, et réplication
de l'ADN viral
L'ADN génomique simple brin est d'abord pris en charge par des enzymes
de la cellule hôte, qui en synthétisant le brin complémentaire
transforment le génome viral en and double brin.
Les séquences répétées inversées (ITR) aux extremités du génome se
replient sur elles-mêmes, de façon à ce que le groupement OH à
l'extremité 3' de l'ADN génomique du virus puisse servir comme amorce
pour la synthèse du brin complémentaire d'ADN.
La forme double-brin de l'ADN viral comprend un promoteur qui dirige
la transcription des gènes viraux par l'ARN polymérase de la cellule
hôte. L'épissage alternatif génère les différents ARNm du virus, codant
pour les deux protéines de capside (VP1 et VP2), et la protéine non-structurale
NS1. Ces trois protéines sont synthétisées dans le cytoplasme, puis
importées dans le noyau.
La protéine NS1 est essentielle pour la réplication du génome viral –
mais elle n'est pas une polymérase à ADN. La fonction de la protéine NS1
est de se fixer spécifiquement à l'ADN viral double brin, et de diriger
la machinerie cellulaire de réplication de l'ADN vers le génome viral.
Cette activité de la protéine NS1 n'est possible que chez des cellules
qui expriment tous les facteurs cellulaires nécessaires pour la synthèse
de l'ADN. Par conséquent, le parvovirus B-19 ne peut se répliquer que
chez des cellules qui sont déjà engagées dans le cycle cellulaire.
La réplication de l'ADN viral produit de multiples copies brin (+) et
brin (-) de l'ADN viral. Les deux types d'ADN viral simple brin peuvent
être:
- convertis en ADN double brin, qui servira ensuite de matrice
pour la transcription des ARNm viraux, et pour la réplication de
l'ADN viral
- incorporés dans des virions
Assemblage et Libération
Les particules virales sont assemblées dans le noyau, et libérées par
lyse cellulaire.
|
A retenir à
propos de la stratégie de réplication des Parvoviridae |
| Ce sont les virus à ADN
avec le génome le plus petit |
| La cellule hôte fournit
la totalité de la machinerie enzymatique pour la
transcription et la maturation des ARNm viraux, et pour la
réplication de l'ADN viral. |
| Les Parvoviridae sont
capables de se répliquer de façon autonome SEULEMENT chez
des cellules qui sont activement en train de se diviser (telles
que les cellules progénitrices des érythrocytes). Dans tout
autre cas, la co-infection par un autre virus à ADN
(Adenovirus ou Herpesvirus) est nécessaire. |
| Les Virus Associés aux
Adenovirus (AAV) sont développés en tant que vecteur de
thérapie génique |
|
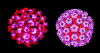 Figure 1. Le virus du papillome © Dr Linda Stannard, Université de Cape Town,
Afrique du Sud. Utilisé avec permission.
Figure 1. Le virus du papillome © Dr Linda Stannard, Université de Cape Town,
Afrique du Sud. Utilisé avec permission. |
Famille
Papillomaviridae
La famille des Papillomaviridae était autrefois regroupés avec la famille
Polyomaviridae dans la famille Papovaviridae (PApillomavirus, POlyomavirus,
et le virus simien VAcuolisant 40) à cause de l’homologie de structure de
leurs capsides. Cependant, il est maintenant clair que les deux groupes de
virus déploient des stratégies de réplication suffisamment différentes pour
justifier leur séparation en deux familles distinctes.
|
Propriétés des Papillomaviridae et des
Polyomaviridae |
| Petite
taille : 40 à 60 nm de diamètre |
| Capside
icosaédrique composée d’une protéine majeure, VP1, et de deux protéines
mineurs, VP2 et VP3 |
| Non-enveloppé |
| Génome en
ADN double brin circulaire, associé avec des histones à l’intérieur de
la particule virale |
Les Papillomaviridae (Figure 1) sont
difficilement cultivables, et les détails de leur cycle de réplication ne seront
pas présentés ici. Par contre l’impact des Papillomaviridae sur la santé est
traité dans le chapitre sur les virus oncogènes à ADN.
|
 Figure 2. Le virus SV40, un Polyomavirus
©
Dr J-Y
Sgro, University of Wisconsin. Used with permission
Figure 2. Le virus SV40, un Polyomavirus
©
Dr J-Y
Sgro, University of Wisconsin. Used with permission |
Famille Polyomaviridae
Il s'agit notamment des virus polyoma, SV40, BK, et JC. Ils sont tous des
virus icosaédriques non enveloppés de petite taille (diamètre ~ 40 nm), qui
se répliquent dans le noyau. Ils emploient tous une stratégie semblable pour
assurer la réplication de leur ADN génomique. En fonction de la cellule hôte,
ils peuvent soit transformer la cellule (aller ici) soit compléter un cycle
de réplication productif (dit cycle lytique) qui aboutit à la lyse de la
cellule infectée et la libération de nouvelles particules virales.
Cycle de réplication
Attachement, entrée et décapsidation
Les protéines de capside virale interagissent avec les récepteurs
glycosidiques à la surface cellulaire et l’entrée s’effectue
vraisemblablement par endocytose. Les particules virales sont
transportées vers le noyau et la décapsidation a lieu à l’entrée du
noyau. L’ADN (et les histones associées) entre dans le noyau,
probablement à travers le pore nucléaire. Aucune protéine virale ne
rentre dans le noyau de la cellule infectée.
Production d'ARNm et des protéines virales
L'expression des gènes des Polyomaviridae est divisée en phases précoces
et tardives.
Les gènes précoces codent pour les enzymes et les protéines régulatrices
nécessaires pour démarrer la réplication de l’ADN virale, et
l’expression des gènes tardifs.
Les gènes tardifs codent pour les protéines de structure et les
protéines nécessaires pour l'assemblage du virus mature.
|
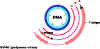 Figure 3 Expression des gènes précoces chez le virus SV40
Figure 3 Expression des gènes précoces chez le virus SV40
Remarque: ---- indique les régions du transcrit primaire qui sont éliminées
lors de l’épissage alternatif des ARNm.
D’après Fiers et al., Nature 273:113

Figure 4. Expression des gènes tardifs
Remarque: ---- indique les régions du transcrit primaire qui sont
éliminées lors de l’épissage alternatif des ARNm. Les flèches larges
indiquent les régions traduites en protéines
D’après Fiers et al., Nature 273:113
|
Phase précoce
du cycle de réplication
Expression des gènes
précoces (figure 3)
Dès l’entrée de l’ADN viral dans le noyau, le promoteur précoce
est reconnu par ARN polymérase II de l'hôte, grâce à un
enhanceur fort dans le génome du SV40. La modification post-transcriptionnelle
d'ARN (l’ajout de la coiffe 5’, la méthylation, la
polyadénylation, l’épissage, etc) est effectuée par les enzymes
de l’hôte. La transcrit précoce primaire subit un épissage
alternatif, ce qui produit deux ARNm distinctes, codant pour les
antigènes "petit t" (tAg) et "grand t" (TAg). Les protéines tAg
et TAg commencent par la même séquence N-terminale, mais leurs
domaines C-terminaux sont différents.
Les ARNm matures sont exportés vers le cytoplasme, puis traduits.
Note: L’utilisation d’un seul transcrit primaire pour coder plus
d’une protéine est un phénomène observé chez plusieurs virus (et
chez la cellule hôte).
Phase tardive du cycle de
réplication
Par définition, la phase tardive commence avec
l'apparition de la réplication du génome viral.
Réplication de l'ADN viral
La réplication de l'ADN du virus SV40/polyoma s’effectue dans le
noyau, et implique l'ADN polymérase de la cellule hôte. Cela
pose deux problèmes au virus SV40 :
-
Une cellule quiescente n’exprime pas toutes
les enzymes nécessaires à la réplication de l’ADN – le virus
doit donc induire l’entrée de la cellule infectée dans le
cycle cellulaire.
-
La fonction normale de l’ADN polymérase
cellulaire est de répliquer l’ADN cellulaire, pas l’ADN
viral. Il faut donc cibler l’ADN polymérase vers l’origine
de réplication du génome viral.
L’antigène T résout ces deux problèmes. D’une
part, la protéine Tag stimule l’entrée de la cellule infectée
dans le cycle cellulaire en se fixant sur les protéines
cellulaires p53 et p105Rb. Cette activité de la protéine Tag
joue ainsi un rôle dans la transformation cellulaire et la
formation de tumeurs.
D’autre part, l’antigène T se fixe sur l’origine
de réplication du génome viral, et induit une modification dans
la structure de l’ADN. L’antigène T recrute ensuite la protéine
cellulaire RpA, et ensemble Tag et RpA déroulent les deux brins
de l’ADN viral et forment un site d’initiation de réplication de
l’ADN qui est reconnu par l’ADN polymérase (et d’autres enzymes
et co-facteurs) cellulaire.
La réplication de l'ADN est bidirectionnelle (il ya deux
fourches de réplication par génome d'ADN circulaire ; la
réplication implique des fragments d'Okazaki, l'ADN ligase,
etc.). Le processus de réplication de l'ADN est très similaire à
celui qui se produit dans la cellule hôte - ce qui n'est guère
surprenant, vu que le virus se sert principalement de la
machinerie moléculaire de l'hôte.
Des histones se complexent avec l'ADN nouvellement synthétisé.
Expression des gènes tardifs
(figure 4)
Les ARNm tardifs sont transcrits après la réplication de l'ADN (une
grande quantité d'ADN viral nouvellement synthétisé est
maintenant disponible en tant que matrice). Les ARNm précoces
sont encore transcrits, mais à un rythme beaucoup plus faible.
L’antigène T est impliqué dans le contrôle de l’augmentation de
transcription à partir du promoteur tardif et la diminution de
la transcription du promoteur précoce.
Les protéines VP1, 2 et 3 sont traduites à partir du même
transcrit primaire qui subit un épissage alternatif (figure 5).
Il en résulte que le cadre de lecture de VP1 est différent de
celui de VP2 et de VP3. Ainsi, une région de l'ADN peut coder
pour les deux séquences d'acides aminés différentes selon quel
cadre de lecture est utilisé. C'est à nouveau un moyen employé
par les virus pour encoder plus d’une protéine sur une courte
séquence unique d'ADN.
Assemblage et libération
Les ARNm VP1, 2 et 3 sont traduits dans le cytoplasme, les
protéines sont transportées vers le noyau, et assemblent
capsides avec l'ADN (histones et cellulaires) à l'intérieur de
la capside. Un grand nombre de capsides s'accumulent dans le
noyau formant un corps d'inclusion. Les virions sont libérés par
lyse cellulaire. La lyse semble être déclenchée par la protéine
VP4, qui est traduite à partir des ARNm VP2 et VP3 par
l’utilisation d’un codon d’initiation en aval de l’AUG utilisé
pour démarrer la traduction de la protéine VP3.
|
 Figure 5 Les protéines VP1, 2, et 3 sont exprimées à partir du même
transcrit primaire, qui subit un épissage alternatif. Le cadre de lecture de
la protéine VP1 n’est pas le même que celui utilisé pour la traduction des
protéines VP2 et VP3.
Figure 5 Les protéines VP1, 2, et 3 sont exprimées à partir du même
transcrit primaire, qui subit un épissage alternatif. Le cadre de lecture de
la protéine VP1 n’est pas le même que celui utilisé pour la traduction des
protéines VP2 et VP3. |
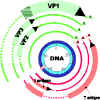 Figure 6 L'expression des gènes chez le virus SV40. Les gènes précoces
sont indiqués en rouge, les gènes tardifs en vert. Note: ---- indique les
régions du transcrit primaire qui sont éliminées lors de l’épissage
alternatif des ARNm. La zone hachurée indique la région de l'ARN traduite
dans deux cadres de lecture différents, en fonction de la nature de l’ARN
épissé qui est traduit. D’après Fiers et al., Nature 273:113
Figure 6 L'expression des gènes chez le virus SV40. Les gènes précoces
sont indiqués en rouge, les gènes tardifs en vert. Note: ---- indique les
régions du transcrit primaire qui sont éliminées lors de l’épissage
alternatif des ARNm. La zone hachurée indique la région de l'ARN traduite
dans deux cadres de lecture différents, en fonction de la nature de l’ARN
épissé qui est traduit. D’après Fiers et al., Nature 273:113 |
|
A retenir à propos de la
stratégie de réplication des Polyomaviridae |
| Fonctions précoces
et tardives |
| Rôle clé de la
protéine multifonctionnelle (TAg) |
| Génome de petite
taille codant pour un nombre limité de protéines |
| Utilisations
multiples d’une séquence d’ADN afin de maximiser le nombre de
protéines encodées sur un petit génome (épissage alternatif, cadres
de lecture chevauchant) |
| La cellule hôte
fournit la machinerie responsable de la synthèse et la maturation
des ARNs viraux, la réplication de l’ADN viral, et les histones
impliqués dans l’empaquetage de l’ADN dans la capside |
|
 Figure 7a Adénovirus visualisé en microscopie électronique. © Dr Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec permission.
Figure 7a Adénovirus visualisé en microscopie électronique. © Dr Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec permission.
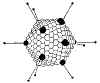 Figure 7b Structure des Adénovirus
Figure 7b Structure des Adénovirus |
Famille Adenoviridae
|
PROPERTIES OF ADENOVIRUSES |
|
Larger than papovaviruses (70nm diameter) |
|
Non-enveloped, icosahedral viruses with fibers at vertices (figure 7 and 8) |
Genome about 7 times size of polyoma virus genome
|
|
The DNA is linear, double stranded, associated with virally
coded, basic proteins in virion (unlike papilloma and polyoma viruses,
adenoviruses do not use cell
histones to package virion DNA) |
|
| |
Cycle de Réplication
Attachement, entrée et décapsidation
Les Adenoviridae infectent généralement les cellules épithéliales. Les
domaines globulaires des fibres de la capside (pIV) se fixent à des
récepteurs à la surface cellulaire, et l’internalisation du virus est
déclenchée ensuite par l’interaction entre des motifs RGD des pentons à
la base des fibres (pIII) avec les intégrines cellulaires. Les
intégrines sont considérés comme des co-récepteurs des adénovirus.
L’acidification de l'endolysosome modifie la structure de la particule
virale, et permet la libération de la protéine pVI de la capside, qui
déstabilise l'endolysosome menant au transfert de la capside virale dans
le cytoplasme. Dans le cytoplasme, la capside est activement transportée
vers le noyau par le réseau des microtubules via une interaction entre
la protéine hexon pII et la dynéine. La decapsidation a lieu à l'entrée
des pores nucléaires, et l’ADN viral rentre ensuite dans le noyau
(figure 9).
|
| |
A

B
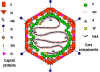 Figure 8 Modèles de la particule virale de l'adénovirus.
Figure 8 Modèles de la particule virale de l'adénovirus.
A: image de reconstruction en 3 dimensions d’une particule intacte d’un
adénovirus vu dans le sens d'un axe de symétrie icosaédrique d’ordre 3 (EMBL
© Virus Structure Resource).
B: Une section stylisée d’une particule d'adénovirus basée sur la
compréhension actuelle de sa composition en protéine et en ADN. Aucune
section réelle de la particule virale ne contiendrait tous les composants
dans le même plan. Les constituants de la particule virale sont désignés par
leur numéro à l'exception de la protéine de terminai (TP). Adapté de Fields
et al., Fundamental Virology (1996).
|
| |
Phase précoce
Expression des gènes précoces
La transcription des gènes précoces des Adenoviridae fait intervenir l’ARN
polymérase de la cellule hôte et permet la transcription des ARNm à partir de
différentes régions du génome virale. Les deux brins peuvent être transcrits
(figure 10). L’utilisation de plusieurs promoteurs différents aboutit à un
contrôle plus flexible de l’expression des gènes précoces. Les ARNm viraux sont
pris en charge par les systèmes cellulaires responsables de l’ajout d’une coiffe
5’ et d’une séquence polyA en 3’. Dans certains cas, les ARNm sont également
d'épissés. Ils sont ensuite exportés vers le cytoplasme et traduits.
Les protéines précoces sont celles qui:
-
sont nécessaires à la transcription des gènes viraux. En particulier, la
protéine E1A est nécessaire pour la transcription des autres gènes précoces. La
protéine E1A est la première protéine virale exprimée par la cellule infectée.
Ainsi le gène E1A est considéré comme un gène "immédiat précoce" tandis que les
autres gènes précoces sont parfois appelés gènes "précoces retardés".
-
sont nécessaires à la synthèse de l'ADN viral (y compris une polymérase à ADN
virale).
-
modifient l'expression de gènes de la cellule hôte. Cela comprend les gènes
dont les produits interférent avec la réponse anti-virale de l'hôte et / ou
interférent avec la régulation du cycle cellulaire. En particulier, la protéine
E1A interagit avec la protéine cellulaire p105-Rb, tandis que la protéine E1B se
fixe sur protéine cellulaire P53, et l’inactive.
|
| |
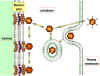 Figure
9 Représentation schématique de l'internalisation et décapsidation des
particules d'adénovirus. Adapté de Zinsser Microbiology 20e éd. Figure
9 Représentation schématique de l'internalisation et décapsidation des
particules d'adénovirus. Adapté de Zinsser Microbiology 20e éd.
 Figure 10
Figure 10
Carte de transcription d'adénovirus. Les gènes précoces sont indiqués en rouge.
Le noir indique les gènes tardifs. Les lignes bleues indiquent l’ADN. Les
crochets montrent les positions des promoteurs. Les transcrits primaires sont
produits à partir de chaque promoteur, puis subissent un épissage alternatif. Le
schéma ci-dessus ne montre pas le transcrit primaire, seulement les régions
présentes dans les produits d'épissage alternatif. Les régions manquantes
indiquent l’élimination d'introns. Adapté du Broker, T.R. Dans Processing of
RNA. (Apirion, D éditeur) 181-212 1984
|
 Figure 11 Réplication de l'ADN d'adénovirus par un mécanisme de
déplacement de brins.
Figure 11 Réplication de l'ADN d'adénovirus par un mécanisme de
déplacement de brins. |
Phase tardive
Réplication de l'ADN
Les Adenoviridae codent pour leur propre polymérase à ADN (qui fait partie des
protéines précoces). L'ADN est répliqué par un mécanisme de déplacement de brin
(figure 11). Il n'y a pas de fragments d'Okazaki, et les deux brins sont
synthétisés de façon continue.
Les polymérases à ADN ne peuvent pas initier la synthèse de novo, ils doivent
prolonger une amorce. Dans le cas de l'adénovirus, la protéine la protéine
virale de terminaison (TP) agit comme une amorce. Il se trouve ainsi liée de
manière covalente à l'extrémité 5 'de tous les brins d'ADN d'adénovirus.
|
|
 Figure
12 Apprêtement d’un transcrit viral primaire Figure
12 Apprêtement d’un transcrit viral primaire |
Transcription tardive
La transcription tardive suit la réplication de l’ADN, mais la façon dont
elle est induite n'est pas bien comprise. Les ARNm tardifs codent
principalement pour des protéines de structure et ils sont transcrits à
partir d’un seul et unique promoteur tardif majeur (figure 10). Le transcrit
primaire généré à partir de ce promoteur est modifié afin de générer divers
ARNm monocistroniques (figures 10 et 12):
Il existe deux types de clivage du transcrit primaire:
On ne sait pas précisément comment ce processus est contrôlé de telle sorte
que les quantités correctes de chaque ARNm soient produits. Cependant, il
semble que le virus produit plus d’ARNm et de protéines virales qu'il n'en
faut pour l’assemblage des particules virales. Il est donc possible qu’un
contrôle de la quantité précise de chaque ARNm synthétisé ne soit pas
nécessaire.
|
| |
Assemblage
L’assemblage de particules d'adénovirus s’effectue dans le noyau. L’ADN
pénètre dans les particules après la formation de capsides immatures. Les
capsides sont alors soumis à un processus de maturation, et ensuite les
cellules infectées sont lysées et les virions s'échappent.
En parallèle, les protéines de structure produites en excès s'accumulent
dans le noyau, où ils forment des corps d'inclusion.
|
| |
|
A retenir à propos de la stratégie de réplication des Adenoviridae
|
| Plus grand et plus
complexe que les Polyomaviridae et les Papillomaviridae |
| Codent pour une
polymérase à ADN virale, et des protéines d’empaquetage d’ADN virales |
| Utilisent l’ARN
polymérase et les systèmes de modification d’ARN de la cellule hôte.
L’expression et la réplication du génome viral doit se faire dans le
noyau. |
| Bien que les
adénovirus disposent de leur propre polymérase à ADN, ils utilisent
plusieurs facteurs de l'hôte pour la réplication de l'ADN. |
|
|
 Figure
13A Structure d’un Herpesvirus Figure
13A Structure d’un Herpesvirus
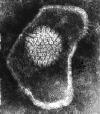 Figure 13B Virus Herpès Simplex visualisé en microscopie électronique.
© Dr
Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec
permission.
Figure 13B Virus Herpès Simplex visualisé en microscopie électronique.
© Dr
Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec
permission.
|
Famille Herpesviridae
|
Propriétés des Herpesviridae |
| Particules virales plus
grandes que les Adenoviridae : 180 à 200 nm de diamètre |
| Génome 3 à 5 fois plus
grand que celui des Adenoviridae |
| Génome en ADN double
brin linéaire |
| Capside icosaédrique
enveloppé. Les Herpesviridae sont donc sensibles aux détergents et aux
solvants. |
|
 Figure 14A Virus Herpès Simplex adsorbé à la membrane plasmique
© Dennis
Kunkel Microscopy, Inc., utilisée avec permission.
Figure 14A Virus Herpès Simplex adsorbé à la membrane plasmique
© Dennis
Kunkel Microscopy, Inc., utilisée avec permission.
 Figure 14B Schématique de la fusion d’un Herpesvirus avec la membrane
plasmique.
Figure 14B Schématique de la fusion d’un Herpesvirus avec la membrane
plasmique. |
Cycle de Réplication
Attachement, entrée et décapsidation
Beaucoup d’Herpesviridae, dont le virus de l'herpès simplex 1 (HSV-1), peuvent
fusionner directement avec la membrane plasmique de la cellule, ce qui permet à
la capside d’entrer dans le cytoplasme de la cellule infectée (figure 14). Une
telle fusion avec la membrane plasmique a des implications à la fois pour le
virus et la cellule hôte. Parmi celles-ci:
- Puisque la protéine de fusion est active à pH neutre, si elle est insérée
dans la membrane de la cellule hôte pendant le cycle de réplication du virus, la
cellule infectée peut potentiellement fusionner avec d'autres cellules et former
des syncytia.
- la membrane virale laisse une "empreinte" dans la membrane plasmique de la
cellule (car les glycoprotéines de l’enveloppe virale se trouveront insérées
dans la membrane plasmique de la cellule infectée), ce qui est un indice que la
cellule est infectée (figure 14b)
Après l’entrée, la capside est transportée vers le noyau. L'ADN viral quitte la
capside via une structure spécialisée située sur l’un des sommets de la capside
virale, le portail viral, qui est orientée spécifiquement vers les pores
nucléaires. Une fois rentrée dans le noyau, l’ADN viral, sous forme linéaire à
l’intérieur de la particule virale, se circularise.
Production d'ARNm et des protéines virales
Les gènes du HSV-1 sont transcrits en trois vagues, dites
α,
β et γ. Les gènes
α et
β sont des gènes précoces, car ils sont transcrits avant la réplication de
l’ADN viral, tandis que les gènes γ sont des gènes tardifs.
|
 Figure
15 Expression des gènes très précoces (α),
précoces (β), et
tardifs (γ) chez le virus Herpès Simplex. Figure
15 Expression des gènes très précoces (α),
précoces (β), et
tardifs (γ) chez le virus Herpès Simplex. |
Phase précoce
La transcription des gènes précoces du HSV-1 est dépendante d’une
polymérase à ARN de la cellule hôte. Toutefois, une protéine de tégument
du virion (VP16), qui est importée dans le noyau lors de l’infection,
joue un rôle essentiel dans l’initiation de la transcription des gènes
α. La protéine VP16 ne se fixe pas directement sur l’ADN viral, mais
elle se fixe sur les protéines Oct-1 cellulaires qui sont recrutées aux
promoteurs α dans le génome viral. Ainsi, la protéine VP16 permet le
recrutement de l'ARN polymérase de l'hôte aux promoteurs
α.
Les gènes
α sont les premiers gènes viraux transcrits, et pour cette
raison les transcrits α sont parfois désignés des transcrits «très
précoces » (Immediate-Early genes). Les enzymes de modification des ARN
prennent en charge les transcrits α, les transformant en ARNm, qui sont
exportés dans le cytoplasme, puis traduits en protéines
α. Les protéines
α sont importées dans le noyau où elles permettent l’activation des
promoteurs β, qui seront utilisés, encore une fois, par l'ARN polymérase
de l'hôte (figure 15).
Les ARNm
β sont également des transcrits précoces car ils sont produits
avant la réplication de l'ADN viral. Parfois le terme gènes «précoce»
fait référence uniquement aux gènes β, parce que l’on utilise le terme «
très précoce » pour décrire les gènes α. Les protéines
β sont impliquées
dans la régulation de l'expression des gènes viraux. Elles diminuent
l'expression des gènes α et elles sont nécessaires pour l'expression des
gènes γ. Elles sont également impliquées dans divers aspects de la
synthèse de l'ADN. Par exemple, les protéines suivantes se trouvent
parmi les protéines β:
- L’ADN polymérase virale
- Des protéines de liaison d'ADN
- La thymidine-kinase virale
- La Ribonucléotide réductase
La thymidine kinase et la ribonucléotide reductase permettent la
production de désoxynucléotides tri-phosphate (dNTP) qui sont les
substrats pour la synthèse de l’ADN. Les dNTPs sont présentes en
quantité très faible dans des cellules quiescentes, ce qui rendrait la
réplication de l’ADN virale peu efficace, voire impossible. Les
Herpesviridae contournent donc cette limitation de la cellule hôte en
produisant la thymidine kinase et la ribonucléotide reductase.
Puisque ces protéines
β sont d'origine virale et ne sont pas des enzymes
de l'hôte, ce sont des maillons potentiellement faibles dans le cycle de
réplication des Herpesviridae, qui peuvent être ciblés par la
chimiothérapie antiviral.
|
|
 Figure 16 Structures génomiques possibles chez les Herpesviridae
Figure 16 Structures génomiques possibles chez les Herpesviridae |
Phase tardive
Réplication de l'ADN viral
Les Herpesviridae codent pour plusieurs protéines, en plus de l'ADN
polymérase, qui sont nécessaires à la réplication de l'ADN viral. Le
mécanisme précis de la réplication de l'ADN ne sera pas présenté ici, mais
elle aboutit à la formation de longues concatémères (des répétitions en
tandem du génome liées tête-à-queue) qui sont clivés en molécules d’ADN à la
taille d’un seul génome lorsque l'ADN est empaqueté dans le virion (figure
16). De plus, la réplication de l'ADN viral est accompagnée d’un fort taux
de recombinaison.
Le génome de plusieurs Herpesviridae (par exemple le HSV-1) montre une
structure caractéristique, dans laquelle deux parties du génome peuvent
s’inverser l’un par rapport à l'autre (figure 16). Chez d’autres
Herpesviridae l’ADN n’est pas organisé de cette façon, et la signification
fonctionnelle de cette structure génomique n’est pas claire.
Transcription tardive:
Par définition, la transcription tardive survient après la réplication de
l'ADN. Les ARNm γ sont transcrits, exportés, puis ils sont traduits dans le
cytoplasme. Les protéines γ sont pour la plupart, des protéines
structurelles.
Au cours du stade tardif, l'expression des gènes
β diminue, probablement dû
à la régulation de la transcription des gènes
β par l'une des protéines γ.
Les gènes α,
β, et γ sont éparpillés un peu partout dans le génome viral –
chez les Herpesviridae il n'y a donc pas d'organisation apparente du génome
en blocs de transcription précoce ou tardive.
|
 Figure 17A Virus Herpès Simplex dans les vacuoles cellulaires et cytoplasme
des lymphocytes du sang périphérique © Dennis Kunkel Microscopy, Inc.,
utilisée avec permission
Figure 17A Virus Herpès Simplex dans les vacuoles cellulaires et cytoplasme
des lymphocytes du sang périphérique © Dennis Kunkel Microscopy, Inc.,
utilisée avec permission
 Figure 17B Virus Herpès Simplex à l’intérieur et à l’extérieur d’un
lymphocyte infecté © Dennis Kunkel Microscopy, Inc., utilisée avec
permission
Figure 17B Virus Herpès Simplex à l’intérieur et à l’extérieur d’un
lymphocyte infecté © Dennis Kunkel Microscopy, Inc., utilisée avec
permission
|
Assemblage
L’assemblage des capsides se produit dans le noyau. Initialement, les
protéines de capside s’associent avec des protéines de charpente virale.
Ensemble, les protéines de capside et les protéines de charpente forment une
structure sphérique, mais qui est pleine, et non pas creuse. Les protéines
de charpente sont alors dégradées par une protéase virale, afin de produire
des capsides vides. L'ADN est alors transféré dans la capside.
Les capsides remplies d’ADN étant trop grosses pour sortir par des pores
nucléaires, les capsides des Herpesviridae quittent le noyau par un autre
moyen. Les capsides acquièrent une enveloppe en sortant du noyau par
bourgeonnement à travers les zones de la membrane nucléaire interne où des
protéines membranaires virales sont insérées (figure 17). Les protéines de
tégument s’accumulent également à la face interne de la membrane nucléaire
interne. L'enveloppe virale fusionne ensuite avec la membrane nucléaire
externe, et la nucléocapside nue est livrée dans le cytoplasme, où il
acquiert un tégument plus mature. Elle est alors enveloppée une deuxième
fois par bourgeonnement dans des vésicules dérivées du Golgi, et finalement
libérée lors de la fusion de ces vésicules de sécrétion avec la membrane
plasmique.
La protéine VP16, essentielle pour la transcription des ARNm
α au prochain tour d'infection fait partie des
protéines de tégument, incorporées dans le virion entre la capside et
l’enveloppe virale.
Pendant l’assemblage des Herpesviridae, des protéines de structure produites
en excès s'accumulent dans le noyau, et forment souvent des corps
d'inclusion, responsables en partie de l'effet cytopathogène du virus.
|
A retenir à propos de
la stratégie de réplication des Herpesviridae |
| Expression des
gènes en trois vagues – ARNm α (très
précoce), β(précoce), et γ (tardif) |
| Les gènes
α, β, et γ
ne sont pas organisés en blocs dans le génome du virus |
| La majorité
des étapes du cycle de réplication sont effectuées par des
protéines virales |
| De ce fait,
les protéines virales impliquées dans la réplication de l’ADN
virale représentent des cibles pharmacologiques |
|
Figure 17C Les étapes de l'exocytose des Herpesviridae
à partir du noyau, dans laquelle la capside est assemblée, jusqu’à la
membrane plasmique.
|
 |
 |
 |
 |
|
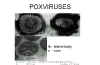 Figure 18 Le virus de la variole en microscopie électronique à contraste
négatif (images d’en haut) et en couche mince (image d’en bas).
© F. Fenner
Figure 18 Le virus de la variole en microscopie électronique à contraste
négatif (images d’en haut) et en couche mince (image d’en bas).
© F. Fenner |
Les virus à ADN cytoplasmique
|
 Figure 19 Garçon avec la variole
Figure 19 Garçon avec la variole
CDC / Cheryl Tryon ctt1@cdc.gov |
Famille Poxviridae Les Poxviridae sont importants pour plusieurs raisons :
- Certains Poxviridae, comme le virus de la variole (figure 19), ou le virus de la
vaccine (utilisé en tant que vaccin contre la variole), ont joué un rôle
important dans l’histoire de la santé publique.
- Les Poxviridae sont des armes biologiques agents potentiels
- Les Poxviridae sont exploités dans le développement de nouveaux vaccins (par
l’utilisation des virus de la vaccine recombinants)
- Certains Poxviridae infectent encore l’Homme (molluscum contagiosum (figure 20),
et les virus « orf », la variole du singe (monkey pox virus), et la vaccine).
- La plupart de nos connaissances viennent de l’observation du virus de la
vaccine, qui est le Poxvirus le plus étudié.
|
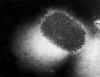 Figure 20 Le poxvirus Molluscum contagiosum en microscopie électronique à
contraste négatif
Figure 20 Le poxvirus Molluscum contagiosum en microscopie électronique à
contraste négatif
CDC |
|
Propriétés des Poxviridae |
| Particules
virales de grande taille – juste visible par microscopie optique |
| Grand
génome en ADN double brin. La taille varie selon le virus, mais les
génomes des Poxviridae sont aussi grands, ou plus grands que ceux des
Herpesviridae |
| Particule
virale enveloppée, de structure complexe. Les particules
extracellulaires possèdent une double enveloppe. |
| |
|
| |
Les Poxviridae se répliquent intégralement dans le cytoplasme. De ce fait, les
Poxviridae doivent fournir leur propre machinerie enzymatique pour la synthèse
des ARNm et pour la réplication de l’ADN viral.
Attachement et Entrée
Le virus se fixe sur des récepteurs à la surface de la cellule hôte, puis rentre
soit par endocytose, soit directement par fusion avec la membrane plasmique.
L’enveloppe est perdue, et le reste de la particule virale rentre alors dans le
cytoplasme.
Phase Précoce
Transcription Précoce
Après l’entrée du virus, un nombre limité d’ARNm « très précoces » sont
transcrits. Evidemment, une transcriptase à ARN dépendante de l’ADN est
nécessaire pour transcrire ces ARNm. Par contre, les polymérases de l’hôte se
trouvent à l’intérieur du noyau, ce qui oblige les Poxviridae à utiliser une
polymérase à ARN dépendante de l’ADN virale. Cette enzyme doit être active
directement après l’entrée du virus. Elle doit donc être incorporée dans la
particule virale afin de rentrer dans la cellule hôte ensemble avec l’ADN viral.
L’ADN génomique purifié d’un Poxvirus n’est pas infectieux, car en l’absence
d’une polymérase à ARN virale aucun ARNm viral, ni protéine virale ne peut être
produit.
Les ARNm des Poxviridae sont coiffés, methylés et polyadenylés, de la même façon
que des ARNm de la cellule hôte. Ces modifications sont également effectuées par
les enzymes virales qui, comme la polymérase à ARN virale, sont incorporées dans
la particule virale. Jusqu’à présent, aucun ARNm épissé n’a été décrit chez les
Poxviridae – ce qui n’est guère étonnant, vu que la « spliceosome » (machinérie
d’épissage) se trouve dans le noyau, tandis que les ARNm des Poxviridae sont
transcrits dans le cytoplasme.
L’un des ARNm « très précoces » code pour une enzyme de décapsidation qui rend
la totalité de l’ADN viral disponible à l’ARN polymérase, et permet la
transcription de tous les gènes « précoces ». L’achèvement de la décapsidation
par une enzyme qui doit être produite de novo chez la cellule infectée est un
caractère unique des Poxviridae.
Les protéines précoces sont impliquées dans la réplication de l’ADN viral, la
transcription et la modification de l’ARN. Un petit nombre de protéines de
structure figurent également parmi les gènes précoces.
|
| |
Phase Tardive
Réplication de l’ADN
Chez les Poxviridae, la réplication de l’ADN dépend entièrement des enzymes
virales. Son mécanisme est inhabituel, et ne sera pas présenté ici. La
réplication de l’ADN se fait uniquement à l’intérieur des « usines » de
production de virus, qui concentrent les protéines et l’ADN viraux. L’ADN
viral n’est pas libéré dans le cytoplasme de la cellule.
Transcription et traduction tardives
Le processus est complexe. Certains protéines tardives sont produites
pendant toute la phase tardive, tandis que d’autres sont exprimées
uniquement au début de la phase tardive. De plus, la production de certaines
protéines précoces se termine dès que la réplication de l’ADN viral se
déclenche, tandis que l’expression d’autres protéines précoces continue
pendant la phase tardive.
La gestion de l’expression des gènes pendant le cycle de réplication des
Poxviridae ne peut donc pas se résumer simplement à un choix entre
l’expression précoce et tardive. Etant donné la taille et la complexité des
génomes des Poxviridae, la complexité du contrôle de l’expression des gènes
chez ces virus n’est guère étonnante.
|
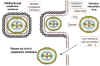 Figure 21 Schéma de la formation et libération des particules infectieuses du
virus la variole. Le core du virus s’enveloppe dans une membrane cytoplasmique
pour former des virions intracellulaires matures (IMV) qui peuvent s'échapper
lors de la lyse de la cellule hôte. En revanche, les virions extracellulaires
enveloppés (EEV) quittent la cellule par bourgeonnement à travers la membrane
plasmique. Les EEV possèdent une double enveloppe. Les IMV et EEV sont
infectieux.
Adapté de Baron, S. Ed. Microbiologie médicale 4e édition. 1996.
Figure 21 Schéma de la formation et libération des particules infectieuses du
virus la variole. Le core du virus s’enveloppe dans une membrane cytoplasmique
pour former des virions intracellulaires matures (IMV) qui peuvent s'échapper
lors de la lyse de la cellule hôte. En revanche, les virions extracellulaires
enveloppés (EEV) quittent la cellule par bourgeonnement à travers la membrane
plasmique. Les EEV possèdent une double enveloppe. Les IMV et EEV sont
infectieux.
Adapté de Baron, S. Ed. Microbiologie médicale 4e édition. 1996. |
Assemblage et Libération
L’assemblage des particules virales a lieu dans le cytoplasme. Les
particules virales immatures acquièrent une enveloppe dans le cytoplasme
– c’est-à-dire en l’absence de bourgeonnement à travers une membrane
cellulaire. Le mécanisme impliqué dans ce processus n’est pas
précisément compris, mais il semble que la capside virale est en quelque
sorte « enrobée » par des membranes cellulaires. Un modèle plus ancien,
proposant que des lipides formant l’enveloppe étaient synthétisées de
novo par le virus à partir de lipides n’est pas exacte.
Les particules enveloppées viennent à maturation progressivement, et le
plus souvent sont libérées par la désintégration de la cellule infectée.
La libération par bourgeonnement est également possible, et dans ce cas,
les particules bourgeonnées possèdent une double enveloppe. Le mécanisme
de libération des particules virales dépend de la nature de la cellule
hôte, et les deux types de particule virale mature sont infectieux.
|
A retenir à propos de la
stratégie de réplication des Poxviridae |
| Réplication
cytoplasmique |
| Grand génome avec
contrôle complexe de l’expression des gènes |
| Transcription et
réplication de l’ADN viral effectuées par des enzymes virales |
| Plusieurs mécanismes
moléculaires uniques au Poxviridae employés à différentes étapes du
cycle de réplication – peu de ressemblance avec des mécanismes
employés par d’autres virus à ADN |
|
|
|
 Retour à la section de virologie de Microbiologie et Immunologie On-line
Retour à la section de virologie de Microbiologie et Immunologie On-line
This page last changed on
Wednesday, November 06, 2013
Page maintained by
Richard Hunt
|

 Figure 1. Le virus du papillome © Dr Linda Stannard, Université de Cape Town,
Afrique du Sud. Utilisé avec permission.
Figure 1. Le virus du papillome © Dr Linda Stannard, Université de Cape Town,
Afrique du Sud. Utilisé avec permission. Figure 2. Le virus SV40, un Polyomavirus
©
Dr J-Y
Sgro, University of Wisconsin. Used with permission
Figure 2. Le virus SV40, un Polyomavirus
©
Dr J-Y
Sgro, University of Wisconsin. Used with permission Figure 5 Les protéines VP1, 2, et 3 sont exprimées à partir du même
transcrit primaire, qui subit un épissage alternatif. Le cadre de lecture de
la protéine VP1 n’est pas le même que celui utilisé pour la traduction des
protéines VP2 et VP3.
Figure 5 Les protéines VP1, 2, et 3 sont exprimées à partir du même
transcrit primaire, qui subit un épissage alternatif. Le cadre de lecture de
la protéine VP1 n’est pas le même que celui utilisé pour la traduction des
protéines VP2 et VP3. Figure 6 L'expression des gènes chez le virus SV40. Les gènes précoces
sont indiqués en rouge, les gènes tardifs en vert. Note: ---- indique les
régions du transcrit primaire qui sont éliminées lors de l’épissage
alternatif des ARNm. La zone hachurée indique la région de l'ARN traduite
dans deux cadres de lecture différents, en fonction de la nature de l’ARN
épissé qui est traduit. D’après Fiers et al., Nature 273:113
Figure 6 L'expression des gènes chez le virus SV40. Les gènes précoces
sont indiqués en rouge, les gènes tardifs en vert. Note: ---- indique les
régions du transcrit primaire qui sont éliminées lors de l’épissage
alternatif des ARNm. La zone hachurée indique la région de l'ARN traduite
dans deux cadres de lecture différents, en fonction de la nature de l’ARN
épissé qui est traduit. D’après Fiers et al., Nature 273:113 Figure 7a Adénovirus visualisé en microscopie électronique. © Dr Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec permission.
Figure 7a Adénovirus visualisé en microscopie électronique. © Dr Linda Stannard, Université de Cape Town, Afrique du Sud. Utilisé avec permission.
 Figure 8 Modèles de la particule virale de l'adénovirus.
Figure 8 Modèles de la particule virale de l'adénovirus.  Figure
9 Représentation schématique de l'internalisation et décapsidation des
particules d'adénovirus. Adapté de Zinsser Microbiology 20e éd.
Figure
9 Représentation schématique de l'internalisation et décapsidation des
particules d'adénovirus. Adapté de Zinsser Microbiology 20e éd.
 Figure 10
Figure 10 Figure 11 Réplication de l'ADN d'adénovirus par un mécanisme de
déplacement de brins.
Figure 11 Réplication de l'ADN d'adénovirus par un mécanisme de
déplacement de brins. Figure 14A Virus Herpès Simplex adsorbé à la membrane plasmique
© Dennis
Kunkel Microscopy, Inc., utilisée avec permission.
Figure 14A Virus Herpès Simplex adsorbé à la membrane plasmique
© Dennis
Kunkel Microscopy, Inc., utilisée avec permission. Figure
15 Expression des gènes très précoces (α),
précoces (β), et
tardifs (γ) chez le virus Herpès Simplex.
Figure
15 Expression des gènes très précoces (α),
précoces (β), et
tardifs (γ) chez le virus Herpès Simplex. Figure 17A Virus Herpès Simplex dans les vacuoles cellulaires et cytoplasme
des lymphocytes du sang périphérique © Dennis Kunkel Microscopy, Inc.,
utilisée avec permission
Figure 17A Virus Herpès Simplex dans les vacuoles cellulaires et cytoplasme
des lymphocytes du sang périphérique © Dennis Kunkel Microscopy, Inc.,
utilisée avec permission



 Figure 18 Le virus de la variole en microscopie électronique à contraste
négatif (images d’en haut) et en couche mince (image d’en bas).
© F. Fenner
Figure 18 Le virus de la variole en microscopie électronique à contraste
négatif (images d’en haut) et en couche mince (image d’en bas).
© F. Fenner Figure 19 Garçon avec la variole
Figure 19 Garçon avec la variole  Figure 20 Le poxvirus Molluscum contagiosum en microscopie électronique à
contraste négatif
Figure 20 Le poxvirus Molluscum contagiosum en microscopie électronique à
contraste négatif  Figure 21 Schéma de la formation et libération des particules infectieuses du
virus la variole. Le core du virus s’enveloppe dans une membrane cytoplasmique
pour former des virions intracellulaires matures (IMV) qui peuvent s'échapper
lors de la lyse de la cellule hôte. En revanche, les virions extracellulaires
enveloppés (EEV) quittent la cellule par bourgeonnement à travers la membrane
plasmique. Les EEV possèdent une double enveloppe. Les IMV et EEV sont
infectieux.
Adapté de Baron, S. Ed. Microbiologie médicale 4e édition. 1996.
Figure 21 Schéma de la formation et libération des particules infectieuses du
virus la variole. Le core du virus s’enveloppe dans une membrane cytoplasmique
pour former des virions intracellulaires matures (IMV) qui peuvent s'échapper
lors de la lyse de la cellule hôte. En revanche, les virions extracellulaires
enveloppés (EEV) quittent la cellule par bourgeonnement à travers la membrane
plasmique. Les EEV possèdent une double enveloppe. Les IMV et EEV sont
infectieux.
Adapté de Baron, S. Ed. Microbiologie médicale 4e édition. 1996.






