| x | x | ||||
|
|
 |
||||
|
x |
BACTERIOLOGÍA | INMUNOLOGÍA | MICOLOGÍA | PARASITOLOGÍA | VIROLOGÍA |
|
|||||
|
|
|||||
|
E-MAIL |
|||||
|
|
|||||
|
PALABRA CLAVE
Aislamiento (cultivo)
Identificación y taxonomía Pruebas bioquímicas (fisiológicas)
Pruebas
moleculares
El perfil químico Detección en ausencia de cultivoReacción en cadena de la polimerasa (PCR) Aglutinación (detección de antígeno) Tinción Serología (detección de anticuerpos) |
Identificación bacteriana en el diagnóstico del laboratorio contra taxonomía El aislamiento e identificación de las bacterias procedentes de pacientes es un apoyo al tratamiento ya que enfermedades infecciosas causadas por bacterias distintas, presentan diferentes cuadros clínicos y consecuencias. Las pruebas de susceptibilidad a antimicrobianos (por ejemplo, el establecimiento de la concentración o mínima inhibitoria o MIC), en aislamientos microbianos particulares, puede ayudar en la selección de los antibióticos útiles en la terapia. El reconocer que ciertas espécies (o cepas) están siendo aislados en forma atípica, puede sugerir que ha ocurrido un brote de enfermedad nosocomial (hospitalaria), por ejemplo, surgido a partir de instrumentos de hospital contaminados o debido a una mala técnica de asepsia, por parte del personal. Cuando se sospecha que un paciente tiene una infección bacteriana, es común que se lleve a cabo el aislamiento de colonias del microorganismo en cultivo puro (observables a simple vista en placas de agar) para luego tipificarlo. La identificación o tipificación se basa en principios taxonómicos básicos aplicados a la microbiología clínica. En el diagnóstico de laboratorio, cada día se deben caracterizar numerosas muestras y los resultados se deben obtener tan pronto como sea posible. Las pruebas deben leerse y realizarse fácilmente y además deben ser de bajo costo. Los métodos clásicos para la tipificación de las bacterias están basados en características morfológicas y metabólicas. Las pruebas diagnósticas se han seleccionado con base en que empíricamente, proveen información suficiente para hacer posible la discriminación entre las especies. Existen numerosas pruebas con resultados diferentes para cada uno de los patógenos objetivo. Adicionalmente, hoy en día las técnicas de biología molecular (basadas en la caracterización de genes específicos o segmentos de ellos) son más comunes en el laboratorio clínico. Los enfoques de la taxonomía moderna a menudo emplean metodologías técnicas más complejas y se interesan por el perfil de la composición estructural de la bacteria, esto involucra aproximaciones basadas en "biología molecular" o "química analítica". Al día de hoy se reconoce que muchos de los esquemas clásicos para la diferenciación de las bacterias muestran muy poco enfoque en sus relaciones genéticas y algunas veces sus resultados son científicamente diferentes. La información mas reciente ha dado como resultado en una re-designación de ciertas especies bacterianas y algunas veces se ha requerido reorganizar totalmente las relaciones entre las familias y aún al interior de muchas familias bacterianas. |
||||
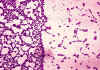
 Figura 1. Cultivos en placas de bicarbonato y agar sangre
de Bacillus anthracis. Colonias lisas en bicarbonato (izquierdo) y
colonias rugosas en el agar sangre (derecho).
CDC/Dr. James Feeley
Figura 1. Cultivos en placas de bicarbonato y agar sangre
de Bacillus anthracis. Colonias lisas en bicarbonato (izquierdo) y
colonias rugosas en el agar sangre (derecho).
CDC/Dr. James Feeley
|
Términos taxonómicos (clasificación) Familia: un grupo de géneros relacionados. Género: un grupo de la especies relacionadas. Especie: un grupo de cepas relacionadas. Tipo: los grupos de cepas dentro de las especies (por ejemplo. biotipos, serotipos). Cepa: aislamiento de una especie en particular. El término más comúnmente utilizado, es el nombre de las especies (por ejemplo. Streptococcus pyogenes - abreviatura S. pyogenes). Hay siempre dos partes del nombre de la especie, uno definirá el género en este caso "Streptococcus" y la otra parte definirá la especie (en este caso "pyogenes"). El nombre del género siempre se escribe con mayúscula y el nombre de la especie no. Ambos, la especie y el género se subrayan o se escriben en itálicas. Pasos para el aislamiento e identificación diagnóstica de bacterias Paso 1. Las muestras de fluidos corporales (por ejemplo, la sangre, la orina, el líquido cerebroespinal) se estrían sobre placas de cultivo y las colonias aisladas de las bacterias (las cuales se observan a simple vista) aparecen después de uno a varios días de incubación (Figura 1). Cada colonia consiste de millones de células bacterianas. La observación de las colonias se caracteriza por su tamaño, la textura, el color y (si ha crecido en agar sangre) las reacciones de hemólisis. La morfología colonial es altamente importante como un primer paso en la identificación bacteriana. En caso de que el organismo requiera oxígeno para el crecimiento, esta otra característica es relevante en la diferenciación. Paso 2. Las colonias se tiñen por la técnica de Gram y las células bacterianas teñidas individualmente se observan bajo el microscopio. Paso 3. Las bacterias se tipifican usando colonias aisladas para sembrar pruebas bioquímicas. Esto a menudo requiere horas adicionales de tiempo de crecimiento. |
||||
|
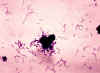
FIGURA 3. EJEMPLOS DE TINCIÓN DE GRAM
|
LA TINCIÓN DE GRAM Un toque del asa sobre la colonia bacteriana se debe disolver sobre una gota de agua y extender como una capa fina sobre un portaobjetos. Se deja secar la preparación, se fija al calor suave y se trata como sigue (Figuras 2 y 3) : Paso 1. Tinción con cristal violeta. Paso 2. Fijación con una solución de yodo. Estabiliza el cristal violeta, todas las bacterias en la preparación permanecen de color púrpura o azul. Paso 3. Extracción con alcohol u otro solvente. Decolora algunas bacterias (las Gram negativas) y no otras (las Gram positivas). Paso 4. Tinción de contraste con safranina. Las bacterias gram positivas ya están teñidas con cristal violeta y permanecen de color púrpura. Las bacterias gram negativas se tiñen de color rosa. A continuación la apariencia de las bacterias se observa bajo el microscopio. Las preguntas que se deben hacer incluyen:
Aparte de la tinción de Gram, hay otras tinciones disponibles que son menos empleadas (por ejemplo tinciones para esporas y cápsulas). De la placa de aislamiento primario se toma otra colonia similar y luego se examinan las propiedades bioquímicas; por ejemplo, ¿fermentara la bacteria un azúcar, por ejemplo la lactosa? Algunas veces, las bacterias se identifican (por ejemplo: por aglutinación) con anticuerpos disponibles comercialmente que reconocen antígenos definidos en la superficie de la bacteria. Ya están disponibles y son actualmente usadas otro tipo de pruebas de diagnóstico molecular.Caracterización taxonómica de bacterias Existe una diversidad considerable aún incluso dentro de las especies. Por lo tanto la comparación de las especies para establecer una separación taxonómica, implica la comparación de múltiples cepas por cada especie en cuestión. Dichas comparaciones están basadas principalmente en análisis químico o molecular.
Análisis
químico
Análisis
molecular
La homología de DNA-DNA (o qué tanto pueden unirse [hibridizar] dos cadenas de DNA de diferentes bacterias) se emplea para comparar las relaciones genéticas de las cepas/especies bacterianas. Si el DNA de dos cepas bacterianas muestra un alto grado de homología (es decir las cadenas se unen muy bien) se considera que estas cepas son miembros de la misma especie. El DNA de especies bacterianas diferentes (a menos que tenga relación taxonómica muy cercana) no muestra homología. En los últimos años, la secuenciación de las moléculas del RNA ribosomal 16S (rRNA 16S) se han convertido en el "estándar de oro" de la taxonomía bacteriana. La molécula tiene aproximadamente mil seiscientos nucleótidos de longitud. La secuencia del rRNA 16S provee una medida de la semejanza genómica, sobre la base de que permite hacer comparaciones de parentesco entre especies de todo el reino bacteriano. Las especies bacterianas cercanamente relacionadas a menudo tienen secuencias de rRNA idénticas. Por lo tanto, esta técnica proporciona información complementaria a la hibridación DNA-DNA. La determinación de la secuencia de los genes de rRNA 16S y otras regiones genéticas se usan en la identificación en el laboratorio de microbiología clínica. Diagnóstico rápido en ausencia de cultivo previo Ciertos patógenos de humanos (incluyendo el agente causal de la tuberculosis, de la enfermedad de Lyme y la sífilis) no pueden ser aislados en el laboratorio o crecen extremadamente poco. Un aislamiento exitoso puede ser lento y en algunos casos imposible. Afortunadamente la detección directa de las bacterias sin cultivarlas es posible en algunos de estos casos. Un acercamiento simple al diagnóstico rápido (como ejemplo: la detección de antígeno) se usa en muchos consultorios médicos para determinación del grupo A de estreptococo. Se toma la muestra con un hisopo a partir de la garganta del paciente y se aglutina el exudado en ausencia de cultivo bacteriano. El antígeno bacteriano se detecta (por aglutinación) con partículas de látex cubiertas con anticuerpos específicos. Las secuencias específicas de DNA se pueden amplificar directamente a partir de fluidos humanos (por medio de la reacción en cadena de la polimerasa, PCR). De esta forma se generan cantidades amplificadas de genes específicos o de porciones de genes y estos pueden ser rápidamente detectados. De esta forma se ha conseguido éxito en el diagnóstico rápido, por ejemplo, de la tuberculosis. Finalmente, la observación directa al microscopio de ciertas muestras para observar la presencia de bacterias puede ser muy útil (por ejemplo, la detección de M. tuberculosis en esputo mediante una tinción para microorganismos ácido-alcohol resistentes). La identificación de la respuesta de anticuerpos en el suero del paciente (perfil serológico) contra el agente infeccioso, solamente puede ser útil después de algunas semanas después de que la infección ha ocurrido.
|
||||
|
RECURSOS DE LA WEB
Tutorial de la tinción de Gram |
|||||
|
SLIDE SHOW |
|||||
|
ANIMACIÓN |
|||||
|
TUTORIAL |
|||||
|
|
Traducido por estudiantes: J. Armando Palacios y Nadia Romero; Profesores: M. en
C. Ana Laura Luna-Torres, Dra. en C. Paula Figueroa-Arredondo
|
||||